Chapter 13 비계층적 군집방법
비계층적 군집방법(Nonhierarchical clustering)은 분할방법(Partitioning method)이라고도 하는데, 군집의 수 \(K\)를 사전에 지정하고 대상 객체들을 적절한 군집에 배정하는 방법이다. 즉, 이 방법은 \(n\)개의 객체를 \(K\)개의 군집에 할당하는 최적화 문제로 간주할 수 있다. 본 장에서는 분할방법의 대표적인 K-means 알고리즘, K-medoids 군집방법, 퍼지 K-means 알고리즘, 그리고 모형기반 군집방법에 대하여 주로 알아본다.
13.1 필요 R package 설치
본 장에서 필요한 R 패키지들은 아래와 같다.
| package | version |
|---|---|
| tidyverse | 1.3.1 |
| stats | 4.1.3 |
| cluster | 2.1.2 |
| flexclust | 1.4-0 |
| mclust | 5.4.7 |
| mvtnorm | 1.1-2 |
13.2 K-means 알고리즘
K-means 알고리즘은 비계층적 군집방법 중 가장 널리 사용되는 것으로 \(K\)개 군집의 중심좌표를 고려하여 각 객체를 가까운 군집에 배정하는 반복적 알고리즘이다.
13.2.1 기본 R 스크립트
10명에 대한 PC의 사용경력(\(x_1\))과 주당 사용시간(\(x_2\))이 다음과 같다.
df <- tribble(
~id, ~x1, ~x2,
1, 6, 14,
2, 8, 13,
3, 14, 6,
4, 11, 8,
5, 15, 7,
6, 7, 15,
7, 13, 6,
8, 5, 4,
9, 3, 3,
10, 3, 2
)
df %>%
knitr::kable(
booktabs = TRUE,
align = c('r', 'r', 'r'),
col.names = c(
'객체번호',
'사용경력($x_1$)', '사용시간($x_2$)'
),
caption = 'PC 사용 데이터'
)| 객체번호 | 사용경력(\(x_1\)) | 사용시간(\(x_2\)) |
|---|---|---|
| 1 | 6 | 14 |
| 2 | 8 | 13 |
| 3 | 14 | 6 |
| 4 | 11 | 8 |
| 5 | 15 | 7 |
| 6 | 7 | 15 |
| 7 | 13 | 6 |
| 8 | 5 | 4 |
| 9 | 3 | 3 |
| 10 | 3 | 2 |
아래와 같이 stats 패키지의 kmeans 함수를 이용하여 K-means 알고리즘 수행 결과를 얻을 수 있다. 아래 스크립트는 군집 수가 \(K = 3\)라 가정하여 수행한 예이다.
set.seed(123)
kmeans_solution <- kmeans(x = df[, -1], centers = 3)위 스크립트 실행 결과 도출된 군집 중심좌표는 위에서 얻어진 kmeans 클래스 객체의 centers값에 저장된다.
kmeans_solution$centers## x1 x2
## 1 13.250000 6.75
## 2 3.666667 3.00
## 3 7.000000 14.00또한 각 학습 데이터가 속한 군집은 cluster값에 저장된다.
kmeans_solution$cluster## [1] 3 3 1 1 1 3 1 2 2 2객체의 군집결과는 아래와 같이 도식화하여 보일 수 있다.
df %>%
mutate(cluster = as.factor(kmeans_solution$cluster)) %>%
ggplot(aes(x = x1, y = x2)) +
geom_text(aes(label = id, color = cluster))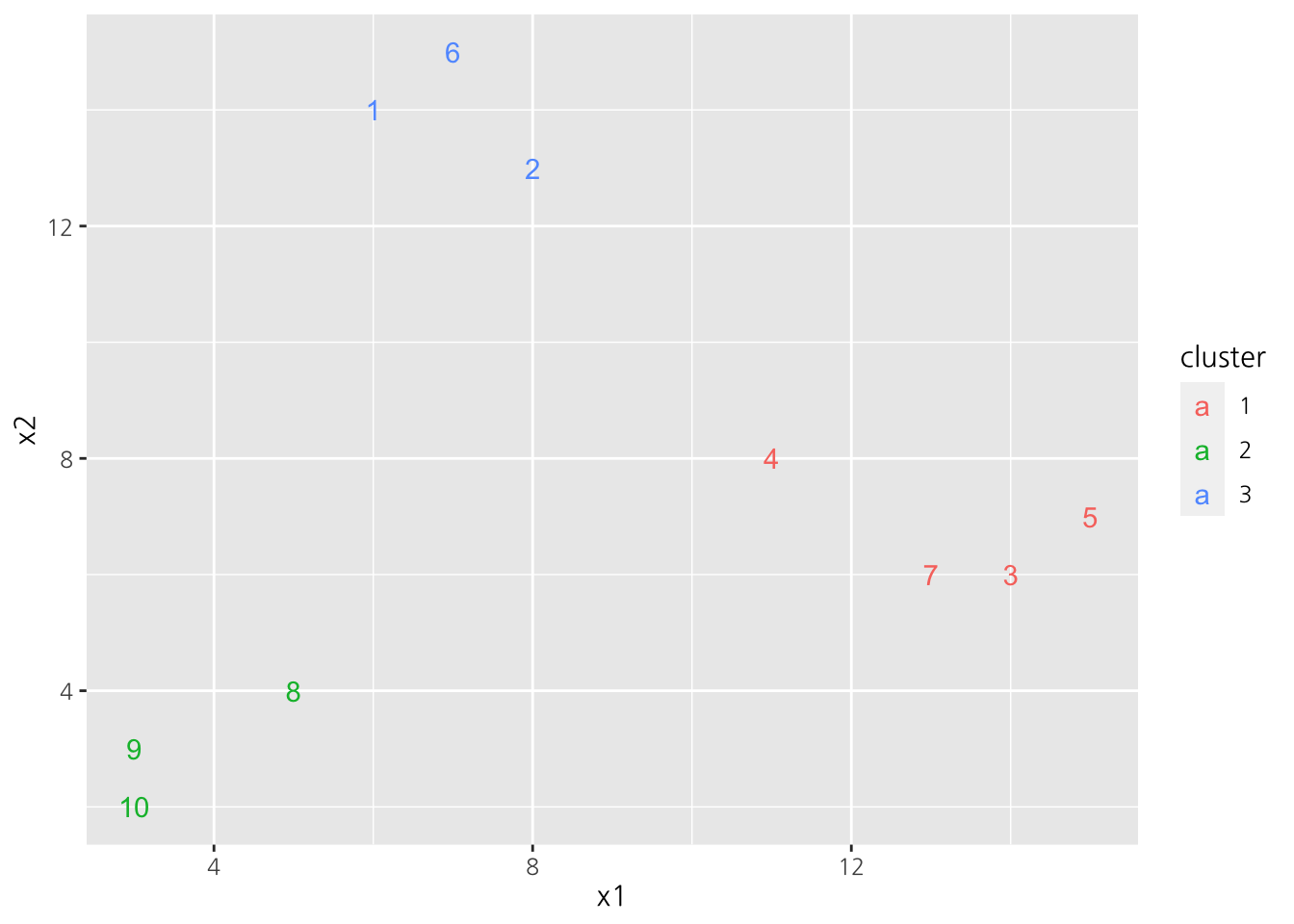
Figure 13.1: K-means 수행 결과
13.2.2 알고리즘
K-means 알고리즘의 구체적 절차는 아래와 같다. Table 13.1에 대한 각 단계의 결과를 함께 살펴보자.
[단계 0] (초기 객체 선정) 어떤 규칙에 의하여 \(K\)개의 객체의 좌표를 초기 군집의 중심좌표(centroid)로 선정한다. 군집 \(j\)의 중심좌표를 \(\mathbf{c}_j = \left(\bar{x}^{(j)}_1, \cdots, \bar{x}^{(j)}_p\right)^\top\)라 하자. 초기 군집 중심좌표 \(\mathbf{c}_1, \cdots, \mathbf{c}_K\)를 선정하는 방법은 예를 들어 다음과 같은 규칙이 사용된다.
- 무작위 방법: 대상 객체 중 무작위로 \(K\)개를 선정한다.
- 외각 객체 선정: 전체 객체의 중심좌표에서 가장 멀리 위치하는 \(K\)개의 객체를 선정한다.
[단계 0] 무작위 방법을 이용하여 Table 13.1으로부터 3개의 객체를 초기 군집 중심좌표로 선정하자.
set.seed(123)
init_cluster <- function(df, k = 1) {
k <- min(k, nrow(df))
df %>%
sample_n(k)
}
cluster_df <- init_cluster(df[, -1], 3)
cluster_df## # A tibble: 3 x 2
## x1 x2
## <dbl> <dbl>
## 1 14 6
## 2 3 2
## 3 8 13[단계 1] (객체의 군집 배정) 각 객체에 대하여 \(K\)개의 군집 중심좌표(centroid)와의 거리(주로 유클리드 거리 사용)를 산출한 후 가장 가까운 군집에 그 객체를 배정한다.
\[\begin{equation*} a_{ij} = \begin{cases} 1 & \text{if } j = \arg\,\max_k d(\mathbf{x}_i, \mathbf{c}_k)\\ 0 & \text{otherwise} \end{cases}, \, i = 1, \cdots, n, \, j = 1, \cdots, K \end{equation*}\]
각 객체에 대하여 3개의 군집 중심좌표와의 거리를 산출해보면 아래와 같다.
flexclust::dist2(df[, -1], cluster_df)## [,1] [,2] [,3]
## [1,] 11.313708 12.369317 2.236068
## [2,] 9.219544 12.083046 0.000000
## [3,] 0.000000 11.704700 9.219544
## [4,] 3.605551 10.000000 5.830952
## [5,] 1.414214 13.000000 9.219544
## [6,] 11.401754 13.601471 2.236068
## [7,] 1.000000 10.770330 8.602325
## [8,] 9.219544 2.828427 9.486833
## [9,] 11.401754 1.000000 11.180340
## [10,] 11.704700 0.000000 12.083046이에 각 객체들에 대해 거리가 가장 가까운 군집에 그 객체를 배정한다.
assign_cluster <- function(df, cluster_df) {
cluster_ind <- flexclust::dist2(df, cluster_df) %>%
apply(1, which.min)
map(unique(cluster_ind), ~which(cluster_ind == .))
}
cluster_objects <- assign_cluster(df[, -1], cluster_df)
cluster_objects## [[1]]
## [1] 1 2 6
##
## [[2]]
## [1] 3 4 5 7
##
## [[3]]
## [1] 8 9 10[단계 2] (군집 중심좌표의 산출) 새로운 군집에 대한 중심좌표를 산출한다.
\[\begin{equation*} \bar{x}^{(j)}_l = \frac{\sum_{i} a_{ij} x_{li}}{\sum_{i} a_{ij}}, \, l = 1, \cdots, p, \, j = 1, \cdots, K \end{equation*}\]
find_center <- function(df, cluster) {
map_dfr(cluster, ~df[., ] %>% summarize_all(mean))
}
new_cluster_df <- find_center(df[, -1], cluster_objects)
new_cluster_df## # A tibble: 3 x 2
## x1 x2
## <dbl> <dbl>
## 1 7 14
## 2 13.2 6.75
## 3 3.67 3[단계 3] (수렴 조건 점검) 새로 산출된 중심좌표값과 이전 좌표값을 비교하여 수렴 조건 내에 들면 마치며, 그렇지 않으면 단계 1을 반복한다.
identical(cluster_df, new_cluster_df)## [1] FALSE위의 경우, 군집 중심좌표가 다르므로 다음 iteration을 진행한다.
13.2.3 R 스크립트 구현
위의 과정을 군집해가 수렴할 때까지 반복하도록 아래와 같이 R 스크립트를 구현해보자.
init_cluster <- function(df, k = 1) {
k <- min(k, nrow(df))
df %>% sample_n(k)
}
assign_cluster <- function(df, cluster_df) {
cluster_ind <- flexclust::dist2(df, cluster_df) %>%
apply(1, which.min)
map(unique(cluster_ind), ~which(cluster_ind == .))
}
find_center <- function(df, cluster) {
map_dfr(cluster, ~df[., ] %>% summarize_all(mean))
}
kmeans_cluster <- function(df, k = 1, verbose = FALSE) {
k <- min(k, nrow(df))
i <- 0L
## 단계 0
cluster_df <- init_cluster(df, k)
while(TRUE) {
i <- i + 1L
## 단계 1
cluster_objects <- assign_cluster(df, cluster_df)
if (verbose) { # 군집해 출력
cat("Iteration", i, ":",
map(cluster_objects, ~ str_c("{", str_c(., collapse = ", "), "}")) %>%
str_c(collapse = ", "),
"\n"
)
}
## 단계 2
new_cluster_df <- find_center(df, cluster_objects)
## 단계 3
if(identical(cluster_df, new_cluster_df)) break
cluster_df <- new_cluster_df
}
res <- list(
cluster_centers = cluster_df,
assgined_objects = cluster_objects,
n_iteration = i
)
return (res)
}set.seed(123)
kmeans_solution <- kmeans_cluster(df[, -1], k = 3, verbose = TRUE)## Iteration 1 : {1, 2, 6}, {3, 4, 5, 7}, {8, 9, 10}
## Iteration 2 : {1, 2, 6}, {3, 4, 5, 7}, {8, 9, 10}위와 같이 2번째 Iteration에서 군집해가 수렴하였으며, 최종 군집해는 아래와 같다.
kmeans_solution$assgined_objects## [[1]]
## [1] 1 2 6
##
## [[2]]
## [1] 3 4 5 7
##
## [[3]]
## [1] 8 9 1013.3 K-medoids 군집방법
K-means 알고리즘에서는 각 군집의 중심좌표(centroid)를 군집 중심으로 고려하고 있는 반면, K-medoids 군집방법에서는 각 군집의 대표객체를 군집 중심으로 고려한다.
K-medoids 군집방법의 알고리즘으로 잘 알려진 것에는 다음과 같은 것들이 있다.
- PAM(Partitioning Around Medoids)
- CLARA(Clustering LARge Applications)
- CLARANS(Clustering Large Applications based on RANdomized Search)
- K-means-like 알고리즘
13.3.1 PAM 알고리즘
PAM 알고리즘은 Kaufman and Rousseeuw (1990) 에 의하여 발표된 것으로, 초기 대표객체를 선정하는 방법인 BUILD와 더 나은 군집해를 찾아나가는 과정인 SWAP의 두 부분으로 구성되어 있다.
13.3.1.1 기본 R 스크립트
df <- tribble(
~id, ~x1, ~x2,
1, 3, 3,
2, 5, 4,
3, 11, 8,
4, 13, 6,
5, 14, 6,
6, 15, 7
)
df %>%
knitr::kable(
booktabs = TRUE,
align = c('r', 'r', 'r'),
col.names = c(
'객체번호',
'사용경력($x_1$)', '사용시간($x_2$)'
),
caption = 'PC 사용자 데이터'
)| 객체번호 | 사용경력(\(x_1\)) | 사용시간(\(x_2\)) |
|---|---|---|
| 1 | 3 | 3 |
| 2 | 5 | 4 |
| 3 | 11 | 8 |
| 4 | 13 | 6 |
| 5 | 14 | 6 |
| 6 | 15 | 7 |
PAM 알고리즘은 cluster 패키지 내의 함수 pam을 이용하여 아래와 같이 간단하게 실행할 수 있다.
pam_solution <- cluster::pam(df[, -1], k = 2)얻어진 pam 객체의 원소 id.med는 몇 번째 객체가 군집의 대표객체로 선정되었는지를 나타낸다.
pam_solution$id.med## [1] 2 5또한 pam 객체의 원소 clustering은 각 객체가 어떠한 군집에 할당되었는지를 보여준다.
pam_solution$clustering## [1] 1 1 2 2 2 213.3.1.2 PAM 알고리즘
PAM 알고리즘의 각 단계를 Table 13.2의 예제 데이터에 적용하여 살펴보기로 하자.
13.3.1.2.1 BUILD
BUILD는 다음 절차들을 거쳐서 \(K\)개의 초기 대표객체를 구하는 과정이다.
[단계 0] 우선 각 객체별로 다른 객체 간의 거리를 구한 후, 그 합이 가장 작은 객체 하나를 대표객체로 선정한다. 선정된 대표객체집합을 \(M\)이라 하자.
pairwise_distance_df <- dist(df[, -1], upper = TRUE) %>%
broom::tidy() %>%
mutate_if(is.factor, ~ as.integer(as.character(.)))
M_idx <- pairwise_distance_df %>%
group_by(item1) %>%
summarize(sum_distance = sum(distance)) %>%
top_n(-1, sum_distance) %>%
.$item1
M <- df[M_idx, ]
M## # A tibble: 1 x 3
## id x1 x2
## <dbl> <dbl> <dbl>
## 1 4 13 6[단계 1] 대표객체로 선정되지 않은 객체 \(j\)에 대하여, 이전에 대표객체로 선정된 객체들 중 객체 \(j\)에 가장 가까운 거리 \(D_j\)를 구한다. 즉,
\[\begin{equation*} D_j = \min_{k \in M} d(j, k), \, j \notin M \end{equation*}\]
D_j <- pairwise_distance_df %>%
filter(
!item1 %in% M$id,
item2 %in% M$id
) %>%
group_by(item1) %>%
top_n(-1, distance) %>%
ungroup() %>%
rename(D_j = distance) %>%
select(-item2)
D_j## # A tibble: 5 x 2
## item1 D_j
## <int> <dbl>
## 1 1 10.4
## 2 2 8.25
## 3 3 2.83
## 4 5 1
## 5 6 2.24그리고 대표객체로 선정되지 않은 두 객체 \(i\), \(j\)에 대하여 다음을 산출한다.
\[\begin{equation*} C_{ji} = \max \left(D_j - d(j, i), 0\right) \end{equation*}\]
d_ji <- pairwise_distance_df %>%
filter(
!item1 %in% M$id,
!item2 %in% M$id
)
C_ji <- D_j %>%
inner_join(d_ji, by = "item1") %>%
mutate(C_ji = pmax(D_j - distance, 0))
C_ji## # A tibble: 20 x 5
## item1 D_j item2 distance C_ji
## <int> <dbl> <int> <dbl> <dbl>
## 1 1 10.4 2 2.24 8.20
## 2 1 10.4 3 9.43 1.01
## 3 1 10.4 5 11.4 0
## 4 1 10.4 6 12.6 0
## 5 2 8.25 1 2.24 6.01
## 6 2 8.25 3 7.21 1.04
## 7 2 8.25 5 9.22 0
## 8 2 8.25 6 10.4 0
## 9 3 2.83 1 9.43 0
## 10 3 2.83 2 7.21 0
## 11 3 2.83 5 3.61 0
## 12 3 2.83 6 4.12 0
## 13 5 1 1 11.4 0
## 14 5 1 2 9.22 0
## 15 5 1 3 3.61 0
## 16 5 1 6 1.41 0
## 17 6 2.24 1 12.6 0
## 18 6 2.24 2 10.4 0
## 19 6 2.24 3 4.12 0
## 20 6 2.24 5 1.41 0.822이는 객체 \(i\)가 추가로 대표객체가 된다고 할 때, 객체 \(j\)의 입장에서 거리 감소량이다.
[단계 2] 다음과 같이 거리감소량이 가장 큰 객체 \(m\)을 대표객체에 포함시키고,
\[\begin{equation*} m = \arg\,\max_{i \notin M} \sum_{j \notin M} C_{ji} \end{equation*}\]
m <- C_ji %>%
group_by(item2) %>%
summarize(sum_C_ji = sum(C_ji)) %>%
top_n(1, sum_C_ji) %>%
.$item2
m## [1] 2대표객체집합을 수정한다.
\[\begin{equation*} M \leftarrow M \cup \{m\} \end{equation*}\]
M <- M %>%
bind_rows(df[m, ])[단계 3] \(K\)개의 대표객체가 선정되었으면 Stop, 그렇지 않은 경우에는 [단계 1]로 되돌아간다.
13.3.1.2.2 SWAP
SWAP은 대표객체로 선정되어 있는 객체 \(i\)와 선정되지 않은 객체 \(h\)를 교환할 때 목적함수의 변화량을 산출해 더 나은 목적함수값을 찾아가는 과정이다.
[단계 1] 객체 \(i\)와 객체 \(h\)를 교환할 때 목적함수의 변화량을 산출하기 위하여 우선, 대표객체로 선정되지 않은 임의의 객체 \(j \neq h\)에서의 변화량을 다음과 같이 산출한다.
\[\begin{eqnarray*} C_{jih} &=& \text{($i$와 $h$를 교환 후 객체 $j$와 대표객체와의 거리)}\\ & & - \text{(교환 전 객체 $j$와 대표객체와의 거리)},\\ & & \, (j \notin M, i \in M, h \notin M) \end{eqnarray*}\]
# 교환 전 각 객체와 대표 객체와의 거리
D_j <- pairwise_distance_df %>%
filter(
!item1 %in% M$id,
item2 %in% M$id
) %>%
group_by(item1) %>%
top_n(-1, distance) %>%
ungroup() %>%
rename(j = item1) %>%
select(-item2)
D_j## # A tibble: 4 x 2
## j distance
## <int> <dbl>
## 1 1 2.24
## 2 3 2.83
## 3 5 1
## 4 6 2.24# 교환할 객체
swap_ids <- tibble(
i = rep(M$id, each = nrow(df) - nrow(M)),
h = rep(setdiff(df$id, M$id), nrow(M))
)
# 교환 후 각 객체와 대표 객체와의 거리
update_distance <- function(i, h, distance_df, center_ids) {
new_center_ids <- c(setdiff(center_ids, i), h)
res <- distance_df %>%
filter(
!item1 %in% c(center_ids, h),
item2 %in% new_center_ids
) %>%
group_by(item1) %>%
top_n(-1, distance) %>%
ungroup() %>%
rename(j = item1) %>%
select(-item2) %>%
mutate(
i = i,
h = h
)
return(res)
}
D_jih <- pmap_dfr(
swap_ids,
update_distance,
distance_df = pairwise_distance_df,
center_ids = M$id
)
D_jih## # A tibble: 24 x 4
## j distance i h
## <int> <dbl> <dbl> <dbl>
## 1 3 7.21 4 1
## 2 5 9.22 4 1
## 3 6 10.4 4 1
## 4 1 2.24 4 3
## 5 5 3.61 4 3
## 6 6 4.12 4 3
## 7 1 2.24 4 5
## 8 3 3.61 4 5
## 9 6 1.41 4 5
## 10 1 2.24 4 6
## # … with 14 more rows# 거리의 변화량
C_jih <- D_j %>%
inner_join(D_jih, by = "j", suffix = c("", "_new")) %>%
mutate(diff_distance = distance_new - distance)
C_jih## # A tibble: 24 x 6
## j distance distance_new i h diff_distance
## <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 2.24 2.24 4 3 0
## 2 1 2.24 2.24 4 5 0
## 3 1 2.24 2.24 4 6 0
## 4 1 2.24 9.43 2 3 7.20
## 5 1 2.24 10.4 2 5 8.20
## 6 1 2.24 10.4 2 6 8.20
## 7 3 2.83 7.21 4 1 4.38
## 8 3 2.83 3.61 4 5 0.777
## 9 3 2.83 4.12 4 6 1.29
## 10 3 2.83 2.83 2 1 0
## # … with 14 more rows[단계 2] 대표객체 \(i\)를 \(h\)로 교환하는 경우 총 변화량은 다음과 같다.
\[\begin{equation*} T_{ih} = \sum_{j} C_{jih} \end{equation*}\]
T_ih <- C_jih %>%
group_by(i, h) %>%
summarize(total_diff_distance = sum(diff_distance))## `summarise()` has grouped output by 'i'. You can override using the `.groups` argument.T_ih## # A tibble: 8 x 3
## # Groups: i [2]
## i h total_diff_distance
## <dbl> <dbl> <dbl>
## 1 2 1 0
## 2 2 3 7.20
## 3 2 5 7.38
## 4 2 6 8.20
## 5 4 1 20.8
## 6 4 3 4.49
## 7 4 5 -0.0447
## 8 4 6 1.71이 때 \(\min_{i, h} T_{ih}\)에 대응하는 객체 \(i^*\)와 \(h^*\)를 찾아 \(T_{i^*h^*} < 0\)이면 교환한 후에 다시 [단계 1]으로 돌아가고, \(T_{i^*h^*} \geq 0\)이면 교환하지 않고 stop.
swap_ih <- T_ih %>%
filter(total_diff_distance < 0) %>%
top_n(-1, total_diff_distance)
swap_ih## # A tibble: 1 x 3
## # Groups: i [1]
## i h total_diff_distance
## <dbl> <dbl> <dbl>
## 1 4 5 -0.0447본 예제의 경우 객체 4 대신 객체 5가 새로운 대표객체로 선택되고, 다시 [단계 1]로 넘어간다.
M <- M %>%
anti_join(df[swap_ih$i, ]) %>%
bind_rows(df[swap_ih$h, ])## Joining, by = c("id", "x1", "x2")M## # A tibble: 2 x 3
## id x1 x2
## <dbl> <dbl> <dbl>
## 1 2 5 4
## 2 5 14 6SWAP 과정이 종료된 후, 최종 군집해는 각 객체를 가장 가까운 대표객체가 속한 군집에 할당함으로써 얻어진다.
13.3.1.3 R 스크립트 구현
일련의 과정을 함수로 구현해보자.
# 각 객체로부터 가장 가까운 대표객체까지의 거리
distance_from_medoids <- function(distance_df, medoids_ids) {
distance_df %>%
filter(
!item1 %in% medoids_ids,
item2 %in% medoids_ids
) %>%
group_by(item1) %>%
arrange(distance) %>%
slice(1) %>%
ungroup() %>%
rename(j = item1) %>%
select(-item2)
}
# k개의 초기 대표객체를 선정
build_medoids <- function(distance_df, k = 1L) {
k <- min(k, length(unique(distance_df$item1)))
# 첫 번째 대표객체 선정
medoids_ids <- distance_df %>%
group_by(item1) %>%
summarize(sum_distance = sum(distance)) %>%
arrange(sum_distance) %>%
slice(1) %>%
.$item1
while (length(medoids_ids) < k) {
D_j <- distance_from_medoids(distance_df, medoids_ids)
d_ji <- distance_df %>%
filter(
!item1 %in% medoids_ids,
!item2 %in% medoids_ids
) %>%
rename(j = item1, i = item2)
# 거리 감소량
C_ji <- D_j %>%
inner_join(d_ji, by = "j",
suffix = c("", "_new")) %>%
mutate(diff_distance = pmax(distance - distance_new, 0))
# 거리 감소량이 가장 큰 객체 선택
m <- C_ji %>%
group_by(i) %>%
summarize(total_diff_distance = sum(diff_distance)) %>%
arrange(desc(total_diff_distance)) %>%
slice(1) %>%
.$i
# 대표객체에 추가
medoids_ids <- c(medoids_ids, m)
}
return(medoids_ids)
}
# 교환 후 각 객체와 대표 객체와의 거리
update_distance <- function(i, h, distance_df, medoids_ids) {
new_medoids_ids <- c(setdiff(medoids_ids, i), h)
res <- distance_from_medoids(distance_df, new_medoids_ids) %>%
filter(j != h) %>%
mutate(
i = i,
h = h
)
return(res)
}
# 교환할 medoid 선택
swap_medoids <- function(distance_df, medoids_ids) {
observation_ids <- unique(distance_df$item1)
# 교환 전 각 객체와 대표 객체와의 거리
D_j <- distance_from_medoids(distance_df, medoids_ids)
# 교환할 객체
swap_ids <- tibble(
i = rep(medoids_ids,
each = length(observation_ids) - length(medoids_ids)),
h = rep(setdiff(observation_ids, medoids_ids),
length(medoids_ids))
)
D_jih <- pmap_dfr(
swap_ids,
update_distance,
distance_df = distance_df,
medoids_ids = medoids_ids
)
# 거리의 변화량
C_jih <- D_j %>%
inner_join(D_jih, by = "j", suffix = c("", "_new")) %>%
mutate(diff_distance = distance_new - distance)
T_ih <- C_jih %>%
group_by(i, h) %>%
summarize(total_diff_distance = sum(diff_distance))
swap_ih <- T_ih %>%
filter(total_diff_distance < 0) %>%
arrange(total_diff_distance) %>%
slice(1)
return(list(remove = swap_ih$i, add = swap_ih$h))
}
# 전체 PAM 알고리즘
pam_medoids <- function(distance_df, k = 1L) {
# BUILD
medoids_ids <- build_medoids(distance_df, k)
k <- length(medoids_ids)
# SWAP
while (TRUE) {
swap_medoids <- swap_medoids(distance_df, medoids_ids)
if (is_empty(swap_medoids$remove)) {
break
} else {
medoids_ids <- c(
setdiff(medoids_ids, swap_medoids$remove),
swap_medoids$add
)
}
}
return (medoids_ids)
}위 구현한 함수를 이용하여 아래와 같이 PAM을 실행해보자.
pairwise_distance_df <- dist(df[, -1], upper = TRUE) %>%
broom::tidy() %>%
mutate_if(is.factor, ~ as.integer(as.character(.)))
medoids_ids <- pam_medoids(pairwise_distance_df, k = 2)## `summarise()` has grouped output by 'i'. You can override using the `.groups` argument.
## `summarise()` has grouped output by 'i'. You can override using the `.groups` argument.df[medoids_ids, ]## # A tibble: 2 x 3
## id x1 x2
## <dbl> <dbl> <dbl>
## 1 2 5 4
## 2 5 14 6위와 같이 2개의 대표객체 2, 5가 선정된다.
최종 군집해는 각 객체를 가장 가까운 대표객체가 속한 군집에 할당함으로써 얻어진다.
assign_cluster <- function(distance_df, medoids_ids) {
distance_df %>%
filter(
!item1 %in% medoids_ids,
item2 %in% medoids_ids
) %>%
group_by(item1) %>%
arrange(distance) %>%
slice(1) %>%
ungroup() %>%
bind_rows(
tibble(
item1 = medoids_ids,
item2 = medoids_ids,
distance = 0
)
) %>%
rename(object = item1) %>%
arrange(object) %>%
group_by(item2) %>%
mutate(cluster = str_c("{", str_c(object, collapse = ", "), "}")) %>%
ungroup() %>%
select(-item2)
}
assign_cluster(pairwise_distance_df, medoids_ids)## # A tibble: 6 x 3
## object distance cluster
## <int> <dbl> <chr>
## 1 1 2.24 {1, 2}
## 2 2 0 {1, 2}
## 3 3 3.61 {3, 4, 5, 6}
## 4 4 1 {3, 4, 5, 6}
## 5 5 0 {3, 4, 5, 6}
## 6 6 1.41 {3, 4, 5, 6}13.3.2 CLARA 알고리즘
PAM 알고리즘은 SWAP 부분에서 모든 가능한 경우를 고려하기 때문에, 전체 객체 수가 많은 경우 계산 시간이 매우 길다는 단점이 있다. 이를 보완하기 위해 CLARA는 적절한 수의 객체를 샘플링한 후 이들에 대해 PAM 알고리즘을 적용하여 중심객체를 선정하는 방법이다. 이러한 샘플링을 여러 번 한 후, 이 중 가장 좋은 결과를 택하는 것인데, 반복수는 5번으로 충분한 것으로 분석되고 있다.
자세한 내용은 교재 (전치혁 2012) 참조
13.3.2.1 기본 R 스크립트
clara_solution <- cluster::clara(df[, -1], k = 2)얻어진 clara 객체의 원소 i.med는 몇 번째 객체가 군집의 대표객체로 선정되었는지를 나타낸다.
clara_solution$i.med## [1] 2 5또한 clara 객체의 원소 clustering은 각 객체가 어떠한 군집에 할당되었는지를 보여준다.
clara_solution$clustering## [1] 1 1 2 2 2 213.3.3 CLARANS 알고리즘
교재 (전치혁 2012) 참조
13.3.4 K-means-like 알고리즘
본 알고리즘은 PAM 알고리즘의 단점을 보완하고자 Park and Jun (2009) 에 의해 제안된 것으로, 대표객체를 반복적으로 수정하는데 K-means 알고리즘의 작동 원리를 모방한 K-medoids 군집 방법이다. 따라서 간단하며 계산 시간이 빠른 것이 장점이라 하겠다. 이 알고리즘은 다음과 같이 3단계로 구성되어 있다. 알고리즘의 각 단계를 Table 13.2의 예제 데이터에 적용하여 살펴보기로 하자.
[단계 1] (초기 대표객체 선정) \(K\)개의 초기 대표객체를 선정하며, 각 객체를 가장 가까운 대표객체에 배정하여 초기 군집해를 얻는다.
객체들 간의 거리 \(d(i, j), \, i, j = 1, \cdots, n\)를 구한다.
pairwise_distance_df <- dist(df[, -1], upper = TRUE) %>%
broom::tidy() %>%
mutate_if(is.factor, ~ as.integer(as.character(.)))각 객체 \(j = 1, \cdots, n\)에 대하여 다음을 산출한다.
\[\begin{equation*} v_j = \sum_{i = 1}^{n} \frac{d(i, j)}{\sum_{k = 1}^{n} d(i, k)} \end{equation*}\]
v_j <- pairwise_distance_df %>%
group_by(item2) %>%
mutate(prop = distance / sum(distance)) %>%
ungroup() %>%
group_by(item1) %>%
summarize(sum_prop = sum(prop)) %>%
rename(j = item1)
v_j## # A tibble: 6 x 2
## j sum_prop
## <int> <dbl>
## 1 1 1.67
## 2 2 1.33
## 3 3 0.781
## 4 4 0.661
## 5 5 0.713
## 6 6 0.849이후 \(v_j\)값들을 오름차순으로 정렬하여 가장 작은 \(K\)개의 값을 초기 대표객체로 선정한다. 본 예에서는 \(K = 2\)로 가정하자.
medoids_ids <- v_j %>%
arrange(sum_prop) %>%
slice(1:2) %>%
.$j
medoids_ids## [1] 4 5이후 객체를 배정하여 군집해를 얻는다. 위에서 정의했던 assign_cluster 함수를 재사용하자.
cluster_solution <- assign_cluster(
pairwise_distance_df,
medoids_ids
)
cluster_solution## # A tibble: 6 x 3
## object distance cluster
## <int> <dbl> <chr>
## 1 1 10.4 {1, 2, 3, 4}
## 2 2 8.25 {1, 2, 3, 4}
## 3 3 2.83 {1, 2, 3, 4}
## 4 4 0 {1, 2, 3, 4}
## 5 5 0 {5, 6}
## 6 6 1.41 {5, 6}[단계 2] (대표객체의 수정) 현재의 군집에 배정된 객체들의 대표객체를 구하여 새로운 대표객체로 삼는다. 새로운 대표객체는 같은 군집에 배정된 다른 객체들로부터의 거리의 합이 최소가 되는 객체이다.
find_medoids <- function(distance_df, ids) {
distance_df %>%
filter(
item1 %in% ids,
item2 %in% ids
) %>%
group_by(item1) %>%
summarize(total_distance = sum(distance)) %>%
arrange(total_distance) %>%
slice(1) %>%
.$item1
}
cluster_objects <- cluster_solution %>%
split(.$cluster) %>%
map(~.$object)
medoids_ids <- map_int(
cluster_objects,
find_medoids,
distance_df = pairwise_distance_df
)
medoids_ids## {1, 2, 3, 4} {5, 6}
## 2 5[단계 3] (객체의 배정) 각 객체를 가장 가까운 대표객체에 배정하여 군집해를 얻는다. 군집해가 이전과 동일하면 Stop하고, 그렇지 않으면 [단계 2]를 반복한다.
new_cluster_solution <- assign_cluster(
pairwise_distance_df,
medoids_ids
)
is_converge <- near(
1,
# clusteval::cluster_similarity(
# as.factor(cluster_solution$cluster),
# as.factor(new_cluster_solution$cluster),
# similarity = "rand"
# )
flexclust::randIndex(
as.factor(cluster_solution$cluster),
as.factor(new_cluster_solution$cluster)
)
)
print(is_converge)## ARI
## FALSE위의 경우 첫 번째 iteration에서 군집해가 수정되었으므로 다음 iteration을 수행한다.
13.3.4.1 R 스크립트 구현
위 일련의 과정들을 수행하는 R 함수 스크립트를 구현해보자.
init_medoids <- function(distance_df, k = 1) {
object_ids <- unique(distance_df$item1)
k <- min(k, length(object_ids))
v_j <- distance_df %>%
group_by(item2) %>%
mutate(prop = distance / sum(distance)) %>%
ungroup() %>%
group_by(item1) %>%
summarize(sum_prop = sum(prop)) %>%
rename(j = item1)
medoids_ids <- v_j %>%
arrange(sum_prop) %>%
slice(1:k) %>%
.$j
return(medoids_ids)
}
assign_cluster <- function(distance_df, medoids_ids) {
distance_df %>%
filter(
!item1 %in% medoids_ids,
item2 %in% medoids_ids
) %>%
group_by(item1) %>%
arrange(distance) %>%
slice(1) %>%
ungroup() %>%
bind_rows(
tibble(
item1 = medoids_ids,
item2 = medoids_ids,
distance = 0
)
) %>%
rename(object = item1) %>%
arrange(object) %>%
group_by(item2) %>%
mutate(cluster = str_c("{", str_c(object, collapse = ", "), "}")) %>%
ungroup() %>%
select(-item2)
}
find_medoids <- function(distance_df, ids) {
distance_df %>%
filter(
item1 %in% ids,
item2 %in% ids
) %>%
group_by(item1) %>%
summarize(total_distance = sum(distance)) %>%
arrange(total_distance) %>%
slice(1) %>%
.$item1
}
kmeans_like_kmedoids <- function(distance_df, k = 1) {
medoids_ids <- init_medoids(distance_df, k)
while (TRUE) {
cluster_solution <- assign_cluster(distance_df, medoids_ids)
cluster_objects <- cluster_solution %>%
split(.$cluster) %>%
map(~.$object)
medoids_ids <- map_int(
cluster_objects,
find_medoids,
distance_df = distance_df
)
new_cluster_solution <- assign_cluster(distance_df, medoids_ids)
is_converge <- near(
1,
flexclust::randIndex(
as.factor(cluster_solution$cluster),
as.factor(new_cluster_solution$cluster)
)
)
if (is_converge) break
}
return(medoids_ids)
}위에서 정의한 함수 kmeans_like_kmedoids를 Table 13.2의 데이터에 적용한 군집결과 및 군집 대표객체는 아래와 같이 얻어진다.
pairwise_distance_df <- dist(df[, -1], upper = TRUE) %>%
broom::tidy() %>%
mutate_if(is.factor, ~ as.integer(as.character(.)))
medoids_ids <- kmeans_like_kmedoids(pairwise_distance_df, k = 2)
medoids_ids## {1, 2} {3, 4, 5, 6}
## 1 513.4 퍼지 K-means 알고리즘
이 방법은 K-means 알고리즘과 유사하나, 하나의 객체가 여러 군집에 속할 가능성을 허용하는 확률 또는 이를 확장한 퍼지(fuzzy) 개념을 도입한 것이다. 객체 \(i\)가 군집 \(j\)에 속할 확률 \(P_{ij}\)를 구하는 문제이다.
13.4.1 기본 R 스크립트
df <- tibble(
id = c(1:10),
x1 = c(6, 8, 14, 11, 15, 7, 13, 5, 3, 3),
x2 = c(14, 13, 6, 8, 7, 15, 6, 4, 3, 2)
)
df %>%
knitr::kable(
booktabs = TRUE,
align = c('r', 'r', 'r'),
col.names = c(
'객체번호',
'사용경력($x_1$)',
'사용시간($x_2$)'
),
caption = 'PC 사용자 데이터'
)| 객체번호 | 사용경력(\(x_1\)) | 사용시간(\(x_2\)) |
|---|---|---|
| 1 | 6 | 14 |
| 2 | 8 | 13 |
| 3 | 14 | 6 |
| 4 | 11 | 8 |
| 5 | 15 | 7 |
| 6 | 7 | 15 |
| 7 | 13 | 6 |
| 8 | 5 | 4 |
| 9 | 3 | 3 |
| 10 | 3 | 2 |
cluster::fanny(df[, -1], k = 3, metric = "SqEuclidean")## Fuzzy Clustering object of class 'fanny' :
## m.ship.expon. 2
## objective 18.37061
## tolerance 1e-15
## iterations 12
## converged 1
## maxit 500
## n 10
## Membership coefficients (in %, rounded):
## [,1] [,2] [,3]
## [1,] 98 1 1
## [2,] 96 3 2
## [3,] 1 99 1
## [4,] 12 80 8
## [5,] 2 96 2
## [6,] 98 1 1
## [7,] 1 99 1
## [8,] 3 3 94
## [9,] 0 0 99
## [10,] 1 1 98
## Fuzzyness coefficients:
## dunn_coeff normalized
## 0.9225653 0.8838480
## Closest hard clustering:
## [1] 1 1 2 2 2 1 2 3 3 3
##
## Available components:
## [1] "membership" "coeff" "memb.exp"
## [4] "clustering" "k.crisp" "objective"
## [7] "convergence" "diss" "call"
## [10] "silinfo" "data"13.4.2 알고리즘
[단계 0] 초기 \(K\)개의 군집을 임의로 결정한다.
\[\begin{equation*} P_{ij} = \begin{cases} 1 & \text{ if object $i$ belongs to cluster $j$}\\ 0 & \text{ otherwise} \end{cases} \end{equation*}\]
init_cluster <- function(df, k = 1) {
k <- min(k, nrow(df))
while (TRUE) {
cluster_ind <- sample.int(k, size = nrow(df), replace = TRUE)
if (length(unique(cluster_ind)) == k) break
}
map_dfc(unique(cluster_ind), ~ as.double(cluster_ind == .))
}
set.seed(4000)
cluster_membership <- init_cluster(df[, -1], k = 3)## New names:
## * NA -> ...1
## * NA -> ...2
## * NA -> ...3cluster_membership## # A tibble: 10 x 3
## ...1 ...2 ...3
## <dbl> <dbl> <dbl>
## 1 1 0 0
## 2 1 0 0
## 3 0 1 0
## 4 1 0 0
## 5 0 0 1
## 6 0 1 0
## 7 0 0 1
## 8 0 1 0
## 9 1 0 0
## 10 1 0 0[단계 1] 각 군집의 중심좌표를 산출한다.
\[\begin{equation*} \mathbf{c}_j = \frac{\sum_{i = 1}^{n} P_{ij}^{m} \mathbf{x}_i}{\sum_{i = 1}^{n} P_{ij}^{m}} \end{equation*}\]
여기에서 상수 \(m\)은 1보다 큰 값을 사용한다.
find_center <- function(df, p, m) {
wt <- p ^ m
df %>%
summarize_all(weighted.mean, w = wt)
}
cluster_df <- map_dfr(cluster_membership, find_center, df = df[, -1], m = 2)
cluster_df## # A tibble: 3 x 2
## x1 x2
## <dbl> <dbl>
## 1 6.2 8
## 2 8.67 8.33
## 3 14 6.5[단계 2] 군집 membership 계수 \(P_{ij}\)를 업데이트한다.
\[\begin{equation*} P_{ij} = \frac{d(\mathbf{x}_i, \mathbf{c}_j)^{-\frac{1}{m - 1}}}{\sum_{a = 1}^{K} d(\mathbf{x}_i, \mathbf{c}_a)^{-\frac{1}{m - 1}}} \end{equation*}\]
여기에서 거리함수 \(d()\)는 제곱 유클리드 거리를 사용한다.
update_membership <- function(df, cluster_df, m) {
distance_mat <- flexclust::dist2(df, cluster_df) ^ 2
p <- distance_mat ^ (-1 / (m - 1)) %>%
`/`(rowSums(.)) %>%
as_tibble(.name_repair = "minimal")
p
}
update_membership(df[, -1], cluster_df, m = 2)## # A tibble: 10 x 3
## `` `` ``
## <dbl> <dbl> <dbl>
## 1 0.451 0.414 0.135
## 2 0.380 0.483 0.137
## 3 0.00381 0.00730 0.989
## 4 0.139 0.576 0.285
## 5 0.0152 0.0285 0.956
## 6 0.406 0.427 0.166
## 7 0.0231 0.0479 0.929
## 8 0.574 0.311 0.115
## 9 0.542 0.315 0.143
## 10 0.508 0.325 0.16613.4.3 R 스크립트 구현
fuzzy_kmeans <- function(df, k = 1, m = 2, max_iter = 1000L, tol = 1e-9) {
k <- min(k, nrow(df))
i <- 0L
cluster_membership <- init_cluster(df, k)
while (i < max_iter) {
i <- i + 1L
cluster_df <- map_dfr(cluster_membership, find_center, df = df, m = m)
new_cluster_membership <- update_membership(df, cluster_df, m)
if (max(abs(cluster_membership - new_cluster_membership)) < tol) break
cluster_membership <- new_cluster_membership
}
res <- list(
n_iteration = i,
center = cluster_df,
membership = cluster_membership %>% as.matrix()
)
return (res)
}set.seed(4000)
fuzzy_kmeans_solution <- fuzzy_kmeans(df[, -1], k = 3, m = 2)## New names:
## * NA -> ...1
## * NA -> ...2
## * NA -> ...3fuzzy_kmeans_solution$n_iteration## [1] 16fuzzy_kmeans_solution$center## # A tibble: 3 x 2
## x1 x2
## <dbl> <dbl>
## 1 3.64 2.98
## 2 7.00 14.0
## 3 13.4 6.63fuzzy_kmeans_solution$membership##
## [1,] 0.007809655 0.983147737 0.009042608
## [2,] 0.015726915 0.957515486 0.026757600
## [3,] 0.006068358 0.006268614 0.987663029
## [4,] 0.078772454 0.120672309 0.800555237
## [5,] 0.017165082 0.022105992 0.960728926
## [6,] 0.006532987 0.984345339 0.009121674
## [7,] 0.005945712 0.005766360 0.988287928
## [8,] 0.939278603 0.026072757 0.034648640
## [9,] 0.993661877 0.002989486 0.003348637
## [10,] 0.981125198 0.008481303 0.010393499fuzzy_kmeans_solution$membership %>%
apply(1, which.max)## [1] 2 2 3 3 3 2 3 1 1 113.5 모형기반 군집방법
모형기반 군집방법(model-based clustering)에서는 각 객체가 혼합분포(mixture)를 따른다고 가정하여 객체의 군집배정변수를 통계적으로 추정하는 것이다.
13.5.1 기본 R script
df <- tibble(
id = c(1:7),
x1 = c(4, 6, 6, 10, 11, 12, 12),
x2 = c(12, 13, 15, 4, 3, 2, 5)
)
df %>%
knitr::kable(
booktabs = TRUE,
align = c('r', 'r', 'r'),
col.names = c(
'객체번호',
'$x_1$',
'$x_2$'
),
caption = '모형기반 군집 학습 데이터'
)| 객체번호 | \(x_1\) | \(x_2\) |
|---|---|---|
| 1 | 4 | 12 |
| 2 | 6 | 13 |
| 3 | 6 | 15 |
| 4 | 10 | 4 |
| 5 | 11 | 3 |
| 6 | 12 | 2 |
| 7 | 12 | 5 |
Table 13.4의 7개의 객체를 두 개의 군집에 배정하는 간단한 R 스크립트는 아래와 같다. 여기에서 mclust::meVII는 각 군집의 분산-공분산 행렬은 서로 다르되, 각 변수의 분산이 동일하며 공분산은 0이라 가정한다.
set.seed(1024)
# 군집 개수
K <- 2
# z_ik 값 초기화
init_z <- matrix(runif(nrow(df) * K), ncol = K) %>%
`/`(apply(., 1, sum))
# EM 알고리즘 - 분산-공분산 행렬: unequal volume (V), spherical(II)
sol <- mclust::meVII(data = df[, -1], z = init_z)
# 군집 사후확률
sol$z## [,1] [,2]
## [1,] 7.596402e-28 1.000000e+00
## [2,] 8.254183e-27 1.000000e+00
## [3,] 9.463816e-36 1.000000e+00
## [4,] 1.000000e+00 6.832084e-20
## [5,] 1.000000e+00 1.473491e-25
## [6,] 1.000000e+00 4.876251e-31
## [7,] 1.000000e+00 1.480388e-20# 혼합분포
sol$parameters## $pro
## [1] 0.5714286 0.4285714
##
## $mean
## [,1] [,2]
## x1 11.25 5.333333
## x2 3.50 13.333333
##
## $variance
## $variance$modelName
## [1] "VII"
##
## $variance$d
## [1] 2
##
## $variance$G
## [1] 2
##
## $variance$sigma
## , , 1
##
## x1 x2
## x1 0.96875 0.00000
## x2 0.00000 0.96875
##
## , , 2
##
## x1 x2
## x1 1.222222 0.000000
## x2 0.000000 1.222222
##
##
## $variance$sigmasq
## [1] 0.968750 1.222222
##
## $variance$scale
## [1] 0.968750 1.222222
##
##
## $Vinv
## NULL13.5.2 EM 알고리즘
우선, 필요한 기호를 다음과 같이 정의하자.
- \(f_k(\mathbf{x} \, | \, \theta_k)\): 군집 \(k\)에 속하는 객체 \(\mathbf{x}\)의 확률밀도함수(\(\theta_k\)는 관련 파라미터)
- \(\tau_k\): 임의의 객체가 군집 \(k\)에 속할 사전확률 (\(\tau_k \geq 0, \, \sum_{k = 1}^{K} \tau = 1\))
이 때, 임의의 객체 \(\mathbf{x}\)는 다음과 같은 혼합 확률밀도함수를 갖는다.
\[\begin{equation*} f(\mathbf{x} \, | \, \boldsymbol\theta) = \sum_{k = 1}^{K} \tau_k f_k(\mathbf{x} \, | \, \theta_k) \end{equation*}\]
본 장에서는 \(f_k\)가 다변량 정규분포를 나타낸다고 가정하자.
추가로, 각 객체가 속하는 군집에 대한 지시변수 \(z_{ik}\)를 아래와 같이 정의하자.
\[\begin{equation*} z_{ik} = \begin{cases} 1 & \text{ if object $i$ belongs to cluster $k$} \\ 0 & \text{ otherwise} \end{cases} \end{equation*}\]
이 \(z_{ik}\) 변수는 실제값이 관측되지 않는 변수이므로, 최우추정법을 이용하여 그 기대값, 즉 객체 \(i\)가 군집 \(k\)에 속할 확률을 추정한다. 보다 자세한 설명은 교재 (전치혁 2012) 참조.
앞의 13.5.1장에서 수행했던 예제를 단계별로 살펴보기로 하자.
[단계 0] \(\hat{z}_{ik}\)를 초기화한다.
set.seed(1024)
# 군집 개수
K <- 2
# z_ik 추정값 초기화
z_hat <- matrix(runif(nrow(df) * K), ncol = K) %>%
`/`(apply(., 1, sum)) %>%
as_tibble() %>%
`names<-`(str_c("C", 1:K))
z_hat## # A tibble: 7 x 2
## C1 C2
## <dbl> <dbl>
## 1 0.406 0.594
## 2 0.623 0.377
## 3 0.372 0.628
## 4 0.956 0.0444
## 5 0.0214 0.979
## 6 0.681 0.319
## 7 0.264 0.736[단계 1] (M-step) \(\hat{z}_{ik}\)를 바탕으로 파라미터를 추정한다.
우선 \(\tau_k\)와 \(\boldsymbol\mu_k\)를 다음과 같이 추정한다.
\[\begin{equation*} \hat{\tau}_k = \frac{\sum_{i = 1}^{n} \hat{z}_{ik}}{n} \end{equation*}\]
tau_hat <- map(z_hat, mean)
tau_hat## $C1
## [1] 0.4746762
##
## $C2
## [1] 0.5253238\[\begin{equation*} \hat{\boldsymbol\mu}_k = \frac{\sum_{i = 1}^{n} \hat{z}_{ik} \mathbf{x}_i}{\sum_{i = 1}^{n} \hat{z}_{ik}} \end{equation*}\]
mu_hat <- map(z_hat, ~colSums(.*df[, -1]) / sum(.))
mu_hat## $C1
## x1 x2
## 8.644042 7.559792
##
## $C2
## x1 x2
## 8.777757 7.853884분산-공분산 행렬 \(\boldsymbol\Sigma_k\)의 추정은 분산-공분산 구조를 어떻게 가정하느냐에 따라 다르다. 우선, 일반적으로 분산-공분산 행렬 \(\boldsymbol\Sigma_k\)는 아래와 같이 decompose할 수 있다 (Banfield and Raftery 1993).
\[\begin{equation*} \boldsymbol\Sigma_k = \lambda_k \mathbf{D}_k \mathbf{A}_k \mathbf{D}_k^\top \end{equation*}\]
여기에서
- \(\lambda_k = \det{\boldsymbol\Sigma_k}^{1 / 2}\); 군집 \(k\)이 크기와 관련
- \(\mathbf{D}_k\): matrix of eigenvectors of \(\boldsymbol\Sigma_k\); 군집 \(k\)의 방향(orientation)과 관련
- \(\mathbf{A}_k\): diagonal matrix s.t. \(\det{\mathbf{A}_k} = 1\); 군집 \(k\)의 형태와 관련
이다. \(\lambda_k\), \(\mathbf{D}_k\) 및 \(\mathbf{A}_k\)에 적용되는 제약조건에 따라 분산-공분산 모형을 정의할 수 있다. 아래는 본 장에서 다룰 세 가지 모형이다.
tribble(
~model, ~sigma,
"VII", "$\\lambda_k \\mathbf{I}$",
"VEI", "$\\lambda_k \\mathbf{A}$",
"VEE", "$\\lambda_k \\mathbf{D} \\mathbf{A} \\mathbf{D}^\\top$"
) %>%
knitr::kable(
booktabs = TRUE,
align = c('c', 'c'),
col.names = c(
'분산-공분산 모형',
'$\\boldsymbol\\Sigma_k$'
),
caption = 'Within-group 분산-공분산 모형'
)| 분산-공분산 모형 | \(\boldsymbol\Sigma_k\) |
|---|---|
| VII | \(\lambda_k \mathbf{I}\) |
| VEI | \(\lambda_k \mathbf{A}\) |
| VEE | \(\lambda_k \mathbf{D} \mathbf{A} \mathbf{D}^\top\) |
즉,
- “VII”
- for all \(k\),\(\mathbf{D}_k = \mathbf{I}\)
- for all \(k\), \(\mathbf{A}_k = \mathbf{I}\)
- “VEI”
- for all \(k\), \(\mathbf{D}_k = \mathbf{I}\)
- for all \(k\), \(\mathbf{A}_k = \mathbf{A}\)
- “VEE”
- for all \(k\), \(\mathbf{D}_k = \mathbf{D}\)
- for all \(k\), \(\mathbf{A}_k = \mathbf{A}\)
Celeux and Govaert (1995) 에 각 분산-공분산 모형에 대한 추정값을 얻는 반복적 알고리즘이 소개되어 있으며, 이는 교재 (전치혁 2012)에도 설명되어 있다.
우선, 각 군집 \(k\) 내에서의 scatter matrix를 아래와 같이 계산한다.
\[\begin{equation*} \mathbf{W}_k = \sum_{i = 1}^{n} \hat{z}_{ik} (\mathbf{x}_i - \hat{\boldsymbol\mu}_k)(\mathbf{x}_i - \hat{\boldsymbol\mu}_k)^\top \end{equation*}\]
p <- ncol(df[, -1])
W <- map(mu_hat,
~pmap_dfc(list(x = df[, -1], mu = .),
function(x, mu) x - mu)) %>%
map(as.matrix, nrow = p, ncol = p) %>%
map2(z_hat, ~ t(.x) %*% (.y * .x))
W## $C1
## x1 x2
## x1 28.22794 -44.46516
## x2 -44.46516 82.36848
##
## $C2
## x1 x2
## x1 37.16942 -53.17491
## x2 -53.17491 92.90912이후 분산-공분산 모형에 따라 아래와 같이 각 군집의 분산-공분산 행렬을 추정한다.
[VII의 경우] 반복 업데이트의 과정 없이 closed-form으로 해가 존재한다.
\[\begin{equation*} \lambda_k = \frac{Tr(\mathbf{W}_k)}{p \sum_{i = 1}^{n} \hat{z}_{ik}} \end{equation*}\]
\[\begin{equation*} \boldsymbol\Sigma_k = \lambda_k \mathbf{I} \end{equation*}\]
이 때 \(p\)는 관측값의 차원수이다 (\(\mathbf{x} \in \mathbb{R}^p\)).
[VEI의 경우]
VEI-0. 행렬 \(\mathbf{B} = \mathbf{I}\)로 초기화한다.
VEI-1. \(\lambda_k\)값을 아래와 같이 계산한다.
\[\begin{equation*} \lambda_k = \frac{Tr(\mathbf{W}_k \mathbf{B}^{-1})}{p \sum_{i = 1}^{n} \hat{z}_{ik}} \end{equation*}\]
VEI-2. 행렬 \(\mathbf{B}\)를 아래와 같이 업데이트한다.
\[\begin{equation*} \mathbf{B} = \frac{diag\left( \sum_{k = 1}^{K} \frac{\mathbf{W}_k}{\lambda_k} \right)}{\left( \det diag\left( \sum_{k = 1}^{K} \frac{\mathbf{W}_k}{\lambda_k} \right) \right)^{1 / p}} \end{equation*}\]
VEI-3. 결과가 수렴하면 종료, 그렇지 않으면 VEI-1로 돌아간다. 최종 수렴한 결과를 통해 각 군집의 분산-공분산 행렬을 아래와 같이 얻는다.
\[\begin{equation*} \boldsymbol\Sigma_k = \lambda_k \mathbf{B} \end{equation*}\]
[VEE의 경우]
VEE-0. 행렬 \(\mathbf{C} = \mathbf{I}\)로 초기화한다.
VEE-1. \(\lambda_k\)값을 아래와 같이 계산한다.
\[\begin{equation*} \lambda_k = \frac{Tr(\mathbf{W}_k \mathbf{C}^{-1})}{p \sum_{i = 1}^{n} \hat{z}_{ik}} \end{equation*}\]
VEE-2. 행렬 \(\mathbf{C}\)를 아래와 같이 업데이트한다.
\[\begin{equation*} \mathbf{C} = \frac{\sum_{k = 1}^{K} \frac{\mathbf{W}_k}{\lambda_k}}{\left( \det \sum_{k = 1}^{K} \frac{\mathbf{W}_k}{\lambda_k} \right)^{1 / p}} \end{equation*}\]
VEE-3. 결과가 수렴하면 종료, 그렇지 않으면 VEE-1로 돌아간다. 최종 수렴한 결과를 통해 각 군집의 분산-공분산 행렬을 아래와 같이 얻는다.
\[\begin{equation*} \boldsymbol\Sigma_k = \lambda_k \mathbf{C} \end{equation*}\]
위 각 분산-공분산 모형을 추정 과정에 대한 설명은, 보다 일반화된 분산-공분산 구조 \(\lambda_k \mathbf{D}_k \mathbf{A}_k \mathbf{D}_k^\top\)을 추정하는 과정을 각 모형에 추가되는 제약에 따라 조금 더 단순하게 표현한 것이다. 보다 자세한 내용은 Celeux and Govaert (1995) 참조.
아래와 같이 군집 내 분산-공분산 행렬을 추정하는 함수 estimate_mixture_cov를 구현해보자. 해당 함수는 3개의 입력변수를 사용한다.
modelName: 분산-공분산 모형 이름. “VII”, “VEI”, “VEE” 중 하나를 선택한다.W:K개의 scatter matrix (\(\mathbf{W}_1, \cdots, \mathbf{W}_K\))를 원소로 지니는 리스트 (list)z: \(z_{ik}\) 값을 지닌 데이터 프레임 (data.frame). \(n\)개의 행과 \(K\)개의 열로 이루어진다. 즉, 행은 각 관측객체를 나타내며, 열은 각 군집을 나타낸다.
estimate_mixture_cov <- function(modelName, W, z) {
K <- ncol(z)
p <- ncol(W[[1]])
# 초기화
D <- map(1:K, ~ diag(1, p))
A <- map(1:K, ~ diag(1, p))
get_lambda <- function(W, z, D, A, p) {
pmap(
list(W, z, D, A),
function(W, z, D, A, p)
sum(diag(W %*% D %*% solve(A) %*% t(D))) / (sum(z) * p),
p = p
)
}
objective_value <- function(W, z, lambda, D, A, p) {
pmap_dbl(
list(W, z, lambda, D, A),
function(W, z, lambda, D, A, p)
sum(diag(W %*% D %*% solve(A) %*% t(D))) / lambda +
p * sum(z) * log(lambda),
p = p
) %>%
sum()
}
i <- 0L
obj <- Inf
while(TRUE) {
i <- i + 1L
lambda <- get_lambda(W, z, D, A, p)
new_obj <- objective_value(W, z, lambda, D, A, p)
if (obj - new_obj < 1e-9) break
if (modelName == "VII") {
} else if (modelName == "VEI") {
B <- map2(W, lambda, ~ diag(.x) / .y) %>%
reduce(`+`) %>%
diag() %>%
`/`(det(.) ^ (1 / p))
A <- map(1:K, ~ B)
} else if (modelName == "VEE") {
C <- map2(W, lambda, ~ .x / .y) %>%
reduce(`+`) %>%
`/`(det(.) ^ (1 / p))
s <- svd(C)
A <- map(1:K, ~ diag(s$d))
D <- map(1:K, ~ s$u)
} else {
stop("Model ", modelName, " is not supported yet.")
}
obj <- new_obj
}
Sigma <- pmap(
list(lambda, D, A),
function(lambda, D, A) lambda * (D %*% A %*% t(D))
)
return (list(
volume = lambda,
shape = A,
orientation = D,
Sigma = Sigma
))
}초기값으로 주어진 \(\hat{z}_{ik}\) 를 이용하여 “VII” 구조의 분산-공분산 행렬을 구해보자.
estimate_mixture_cov("VII", W, z_hat)## $volume
## $volume$C1
## [1] 16.64239
##
## $volume$C2
## [1] 17.68685
##
##
## $shape
## $shape[[1]]
## [,1] [,2]
## [1,] 1 0
## [2,] 0 1
##
## $shape[[2]]
## [,1] [,2]
## [1,] 1 0
## [2,] 0 1
##
##
## $orientation
## $orientation[[1]]
## [,1] [,2]
## [1,] 1 0
## [2,] 0 1
##
## $orientation[[2]]
## [,1] [,2]
## [1,] 1 0
## [2,] 0 1
##
##
## $Sigma
## $Sigma$C1
## [,1] [,2]
## [1,] 16.64239 0.00000
## [2,] 0.00000 16.64239
##
## $Sigma$C2
## [,1] [,2]
## [1,] 17.68685 0.00000
## [2,] 0.00000 17.68685위 estimate_mixture_cov 함수에서 “VII”보다 일반화된 “VEI”와 “VEE” 분산-공분산 모형에 대한 추정도 구현되어 있다.
# estimate_mixture_cov("VEI", W, z_hat)
# estimate_mixture_cov("VEE", W, z_hat)위 일련의 파리미터 추정을 하나의 함수 GMM_Mstep으로 아래와 같이 구성해보자.
GMM_Mstep <- function(df, z, modelName = "VII") {
tau <- map(z, mean)
mu <- map(z, ~colSums(. * df) / sum(.))
p <- ncol(df)
W <- map(mu, ~pmap_dfc(
list(x = df, mu = .), function(x, mu) x - mu)) %>%
map(as.matrix, nrow = p, ncol = p) %>%
map2(z, ~ t(.x) %*% (.y * .x))
var_cov <- estimate_mixture_cov(modelName, W, z)
res <- list(
tau = tau,
mu = mu,
Sigma = var_cov$Sigma
)
return (res)
}
Mstep_res <- GMM_Mstep(df[, -1], z_hat)
Mstep_res## $tau
## $tau$C1
## [1] 0.4746762
##
## $tau$C2
## [1] 0.5253238
##
##
## $mu
## $mu$C1
## x1 x2
## 8.644042 7.559792
##
## $mu$C2
## x1 x2
## 8.777757 7.853884
##
##
## $Sigma
## $Sigma$C1
## [,1] [,2]
## [1,] 16.64239 0.00000
## [2,] 0.00000 16.64239
##
## $Sigma$C2
## [,1] [,2]
## [1,] 17.68685 0.00000
## [2,] 0.00000 17.68685[단계 2] (E-step) M-step에서의 파라미터 추정치를 바탕으로 \(\hat{z}_{ik}\)를 산출한다.
\[\begin{equation*} \hat{z}_{ik} = \frac{\hat{\tau}_k f_k(\mathbf{x}_i \, | \, \hat{\boldsymbol{\mu}}_k, \hat{\boldsymbol{\Sigma}}_k)}{\sum_{l = 1}^{K} \hat{\tau}_l f_l(\mathbf{x}_i \, | \, \hat{\boldsymbol{\mu}}_l, \hat{\boldsymbol{\Sigma}}_l)} \end{equation*}\]
GMM_Estep <- function(df, tau, mu, Sigma) {
pmap_dfc(list(tau = tau, mu = mu, Sigma = Sigma),
function(df, tau, mu, Sigma)
tau * mvtnorm::dmvnorm(df, mean = mu, sigma = Sigma),
df = df) %>%
`/`(rowSums(.))
}
new_z_hat <- GMM_Estep(df[, -1], Mstep_res$tau, Mstep_res$mu, Mstep_res$Sigma)
new_z_hat## C1 C2
## 1 0.4626878 0.5373122
## 2 0.4568716 0.5431284
## 3 0.4373551 0.5626449
## 4 0.4964082 0.5035918
## 5 0.4934276 0.5065724
## 6 0.4886741 0.5113259
## 7 0.4870085 0.5129915[단계 3] 수렴조건을 만족하면 stop, 그렇지 않으면 [단계 1]을 반복한다. 일반적으로는 우도함수값의 변화량을 수렴조건으로 사용하나, 본 장에서는 간단하게 \(z_{ik}\)값의 변화량을 기준으로 수렴을 판단하도록 하자.
max(abs(z_hat - new_z_hat)) < 1e-9## [1] FALSE위 수식의 값이 TRUE이면 수렴, FALSE이면 [단계 1]을 반복한다.
13.5.3 R 스크립트 구현
위 일련의 과정들을 포함하는 하나의 함수 GMM_EM을 아래와 같이 구현해보자. 앞에서 정의했던 함수 GMM_Mstep 및 GMM_Estep을 재사용한다.
GMM_EM <- function(df, K, modelName = "VII", tol = 1e-9) {
K <- min(K, nrow(df))
i <- 0L
# [단계 0] z_ik 추정값 초기화
z_hat <- matrix(runif(nrow(df) * K), ncol = K) %>%
`/`(apply(., 1, sum)) %>%
as_tibble() %>%
`names<-`(str_c("C", 1:K))
while (TRUE) {
i <- i + 1L
# [단계 1] M-step
Mstep_res <- GMM_Mstep(df, z_hat, modelName)
# [단계 2] E-step
new_z_hat <- GMM_Estep(df, Mstep_res$tau, Mstep_res$mu, Mstep_res$Sigma)
# [단계 3] 수렴조건 확인
if (max(abs(z_hat - new_z_hat)) < tol) break
z_hat <- new_z_hat
}
return (list(z = z_hat,
parameters = Mstep_res,
n_iteration = i))
}위 함수를 학습데이터 Table 13.4에 적용한 결과는 아래와 같다.
VII_res <- GMM_EM(df[, -1], 2, "VII")
# 군집 멤버쉽
VII_res$z## C1 C2
## 1 1.000000e+00 6.786307e-28
## 2 1.000000e+00 8.771316e-27
## 3 1.000000e+00 8.895007e-36
## 4 5.251505e-17 1.000000e+00
## 5 7.046079e-22 1.000000e+00
## 6 1.843399e-26 1.000000e+00
## 7 1.544788e-17 1.000000e+00# 혼합분포
VII_res$parameters## $tau
## $tau$C1
## [1] 0.4285714
##
## $tau$C2
## [1] 0.5714286
##
##
## $mu
## $mu$C1
## x1 x2
## 5.333333 13.333333
##
## $mu$C2
## x1 x2
## 11.25 3.50
##
##
## $Sigma
## $Sigma$C1
## [,1] [,2]
## [1,] 1.222222 0.000000
## [2,] 0.000000 1.222222
##
## $Sigma$C2
## [,1] [,2]
## [1,] 0.96875 0.00000
## [2,] 0.00000 0.9687513.5.4 R 패키지 내 모형기반 군집분석
R 패키지 mclust를 통해, 위에서 살펴본 VII, VEI, VEE 외에 보다 다양한 분산-공분산 모형을 가정한 군집분석을 수행할 수 있다 (Scrucca et al. 2016). 다음은 “EEI” 구조에 대한 수행 예이다.
res_mclust_EEI <- mclust::meEEI(df[, -1], z_hat)위 수행 결과 객체는 리스트 형태이며, 그 원소 중 z는 \(\hat{z}_{ik}\)값을, parameters는 혼합분포 파라미터 추정값 (\(\hat{\tau}_k\), \(\hat{\boldsymbol\mu}_k\), \(\hat{\boldsymbol\Sigma}_k\))을 나타낸다.
res_mclust_EEI$z## [,1] [,2]
## [1,] 6.198742e-26 1.000000e+00
## [2,] 2.193775e-22 1.000000e+00
## [3,] 1.432979e-28 1.000000e+00
## [4,] 1.000000e+00 3.497777e-20
## [5,] 1.000000e+00 1.350932e-26
## [6,] 1.000000e+00 5.217649e-33
## [7,] 1.000000e+00 9.883335e-24res_mclust_EEI$parameters## $pro
## [1] 0.5714286 0.4285714
##
## $mean
## [,1] [,2]
## x1 11.25 5.333333
## x2 3.50 13.333333
##
## $variance
## $variance$modelName
## [1] "EEI"
##
## $variance$d
## [1] 2
##
## $variance$G
## [1] 2
##
## $variance$sigma
## , , 1
##
## x1 x2
## x1 0.7738095 0.000000
## x2 0.0000000 1.380952
##
## , , 2
##
## x1 x2
## x1 0.7738095 0.000000
## x2 0.0000000 1.380952
##
##
## $variance$Sigma
## x1 x2
## x1 0.7738095 0.000000
## x2 0.0000000 1.380952
##
## $variance$scale
## [1] 1.033728
##
## $variance$shape
## [1] 0.7485618 1.3358950
##
##
## $Vinv
## NULLmclust 패키지 내의 함수 densityMclust는 Bayesian information criterion (BIC)을 기준으로 최적의 혼합분포를 찾는 함수이다. 즉, 내부적으로 여러가지 분산-공분산 모형과 군집 수의 조합에 대한 군집분석을 수행한 뒤, 각 조합의 최종결과에서 얻어진 BIC 값을 기준으로 최적의 조합을 선정한다.
res_mclust_opt <- mclust::densityMclust(df[, -1], verbose = FALSE)함수 수행결과 객체의 BIC 원소가 나타내는 결과는 아래와 같다.
res_mclust_opt$BIC## Bayesian Information Criterion (BIC):
## EII VII EEI VEI EVI
## 1 -85.40006 -85.40006 -85.70851 -85.70851 -85.70851
## 2 -62.00992 -63.86240 -63.37677 -65.22485 -65.32204
## 3 -65.47796 NA -66.01911 NA NA
## 4 -69.04346 NA -70.17240 NA NA
## 5 -72.02226 NA -73.96817 NA NA
## 6 -62.27998 NA -64.22589 NA NA
## 7 NA NA NA NA NA
## VVI EEE VEE EVE VVE
## 1 -85.70851 -75.27433 -75.27433 -75.27433 -75.27433
## 2 -67.17013 -64.66516 -66.60332 -65.06811 -66.92481
## 3 NA -67.87781 NA NA NA
## 4 NA -67.31282 NA NA NA
## 5 NA NA NA NA NA
## 6 NA NA NA NA NA
## 7 NA NA NA NA NA
## EEV VEV EVV VVV
## 1 -75.27433 -75.27433 -75.27433 -75.27433
## 2 -65.42506 -67.34154 -66.55375 -68.44666
## 3 -71.48895 NA NA NA
## 4 NA NA NA NA
## 5 NA NA NA NA
## 6 NA NA NA NA
## 7 NA NA NA NA
##
## Top 3 models based on the BIC criterion:
## EII,2 EII,6 EEI,2
## -62.00992 -62.27998 -63.37677수행 결과 가장 큰 BIC 값을 지닌 최적의 조합은 EII 분산-공분산 모형으로 2개의 군집을 가정했을 때 얻어진다.
densityMclust 함수 수행 결과 얻어진 혼합분포를 plotting해보자.
plot(res_mclust_opt, what = "density",
data = df[, -1], points.cex = 0.5)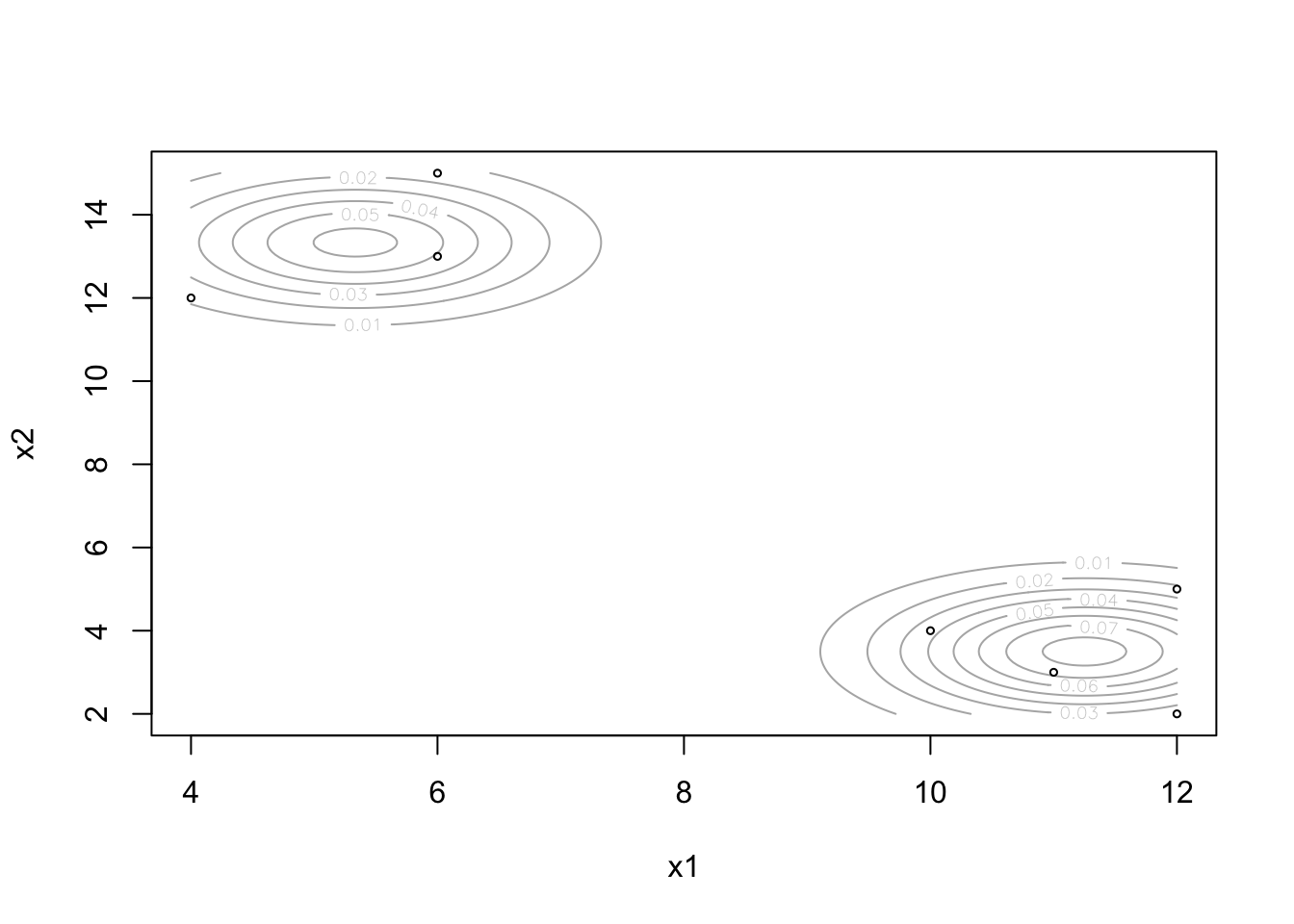
Figure 13.2: mclust::densityMclust 수행 결과 얻어진 혼합분포