Chapter 8 트리기반 기법
8.1 CART 개요
CART(Classification and Regression Trees)는 Breiman et al. (1984) 에 의하여 개발된 것인데, 각 (독립)변수를 이분화(binary split)하는 과정을 반복하여 트리 형태를 형성함으로써 분류(종속변수가 범주형일 때) 또는 회귀분석(종속변수가 연속형일 때)을 수행하는 것이다. 이 때 독립변수들은 범주형 또는 연속형 모두에 적용될 수 있다. 본 장에서는 분류를 위한 목적만을 설명하도록 한다.
8.2 필요 R package 설치
본 장에서 필요한 R 패키지들은 아래와 같다.
| package | version |
|---|---|
| tidyverse | 1.3.1 |
| rpart | 4.1-15 |
| rpart.plot | 3.0.9 |
8.3 CART 트리 생성
8.3.1 기본 R 스크립트
train_df <- tibble(
x1 = c(1,2,2,2,2,3,4,4,4,5),
x2 = c(4,6,5,4,3,6,6,5,4,3),
class = as.factor(c(1,1,1,2,2,1,1,2,2,2))
)| x1 | x2 | class |
|---|---|---|
| 1 | 4 | 1 |
| 2 | 6 | 1 |
| 2 | 5 | 1 |
| 2 | 4 | 2 |
| 2 | 3 | 2 |
| 3 | 6 | 1 |
| 4 | 6 | 1 |
| 4 | 5 | 2 |
| 4 | 4 | 2 |
| 5 | 3 | 2 |
Table 8.1와 같이 두 독립변수 x1, x2와 이분형 종속변수 class의 관측값으로 이루어진 10개의 학습표본을 train_df라는 data frame에 저장한다.
library(rpart)
library(rpart.plot)
cart.est <- rpart(
class ~ x1 + x2
, data = train_df
, method = "class"
, parms = list(split = "gini")
, control = rpart.control(minsplit = 2
, minbucket = 1
, cp = 0
, xval = 0
, maxcompete = 0)
)
rpart.plot(cart.est)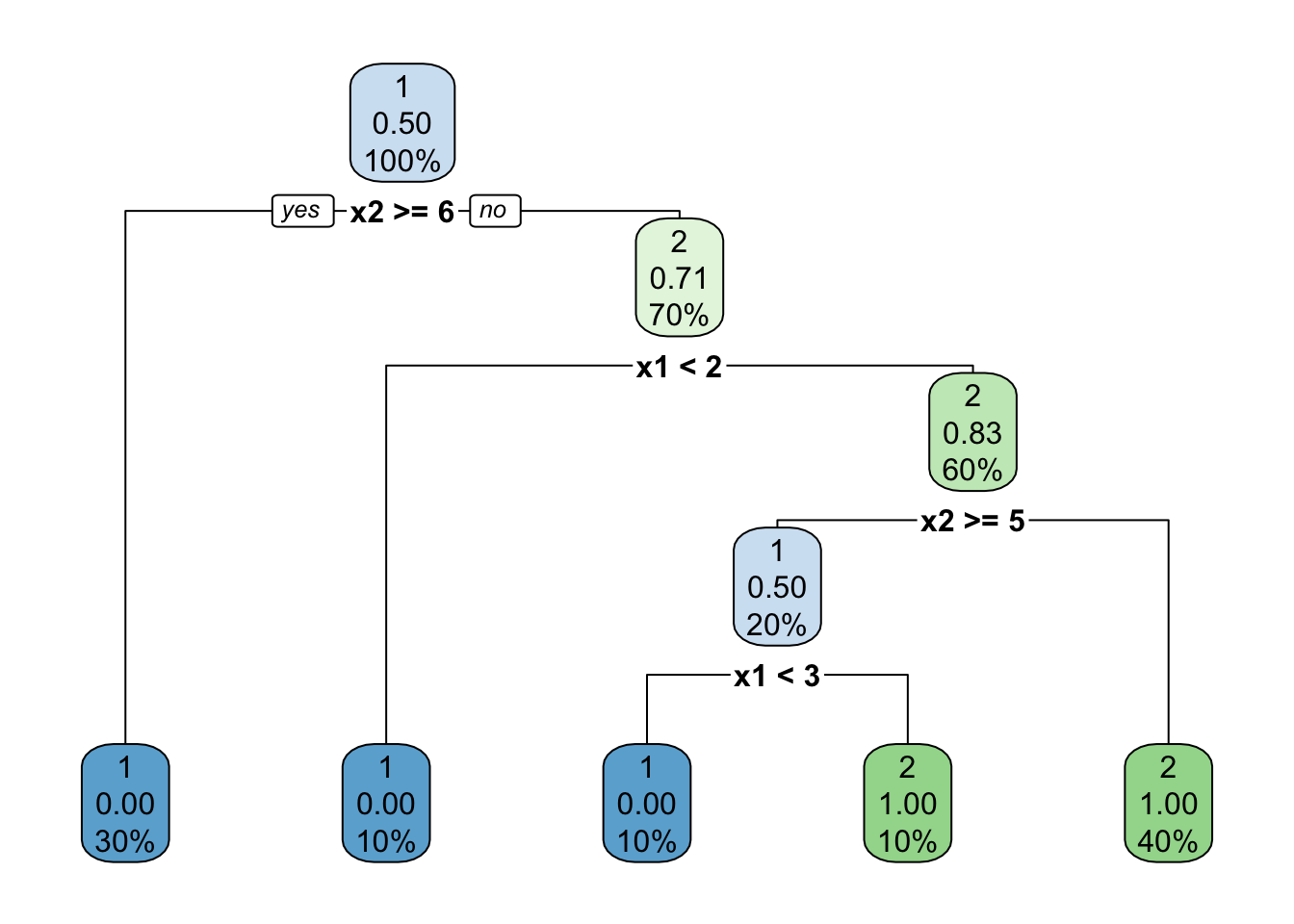
Figure 8.1: CART 트리
rpart 라는 package를 기반으로, 두 변수 x1과 x2를 이용하여 이분형 종속변수 class를 분류하는 CART 트리를 생성할 수 있으며, rpart.plot package를 이용하여 Figure 8.1과 같이 시각화할 수 있다.
8.3.2 기호 정의
본 장에서 사용될 수학적 기호는 아래와 같다.
- \(T\): 트리
- \(A(T)\): 트리 \(T\)의 최종노드의 집합
- \(J\): 범주수
- \(N\): 학습표본의 총 객체수
- \(N_j\): 범주 \(j\)에 속한 객체 수
- \(N(t)\): 노드 \(t\)에서의 객체수
- \(N_j(t)\): 노드 \(t\)에서 범주 \(j\)에 속한 객체수
- \(p(j,t)\): 임의의 객체가 범주 \(j\)와 노드 \(t\)에 속할 확률
- \(p(t)\): 임의의 객체가 노드 \(t\)에 속할 확률 \[p(t) = \sum_{j=1}^{J} p(j,t)\]
- \(p(j|t)\): 임의의 객체가 노드 \(t\)에 속할 때 범주 \(j\)에 속할 조건부 확률 \[p(j|t) = \frac{p(j,t)}{p(t)}, \quad \sum_{j=1}^{J} p(j|t) = 1\]
이 때, 각 확률은 학습표본에서 아래와 같이 추정할 수 있다. \[\begin{align} p(j,t) &\approx \frac{N_j(t)}{N}\\ p(t) &\approx \frac{N(t)}{N}\\ p(j|t) &\approx \frac{N_j(t)}{N(t)} \end{align}\]
8.3.3 노드 및 트리의 불순도
8.3.3.1 노드의 불순도
CART는 지니 지수(Gini index)를 불순도 함수로 사용한다. 총 \(J\)개의 범주별 객체비율을 \(p_1, \cdots , p_J\)라 할 때 (\(\sum_{j=1}^{J} p_j = 1\)), 지니 지수는 식 (8.1)와 같다.
\[\begin{equation} G(p_1, \cdots, p_J) = \sum_{j=1}^{J} p_j(1-p_j) = 1 - \sum_{j=1}^{J}p_j^2 \tag{8.1} \end{equation}\]
노드 \(t\)에서의 범주별 객체비율은 \(p(1|t), \cdots, p(J|t)\)이므로, 노드 \(t\)의 불순도는 식 (8.2)와 같이 산출된다.
\[\begin{equation} \begin{split} i(t) &= 1 - \sum_{j=1}^{J} p(j|t)^2\\ &\approx 1 - \sum_{j=1}^{J} \left[\frac{N_j(t)}{N(t)}\right]^2 \end{split} \tag{8.2} \end{equation}\]
8.3.3.2 트리 불순도
트리 \(T\)의 불순도는 식 (8.3)와 같이 최종노드들의 불순도의 가중평균으로 정의된다.
\[\begin{equation} I(T) = \sum_{t \in A(T)} i(t)p(t) \tag{8.3} \end{equation}\]
여기서 \[ I(t) = i(t)p(t) \] 라 하면, 다음이 성립한다. \[ I(T) = \sum_{t \in A(T)} I(t) \]
8.3.4 분지기준
뿌리 노드에서의 분지만을 살펴보기 위해 control parameter maxdepth의 값을 1으로 설정한다. 이 경우, CART 알고리즘은 뿌리노드에서의 양 갈래 분지만을 선택한 뒤 종료된다. 아래 스크립트를 이용하여 뿌리노드에서 최적분지된 트리를 얻는다.
cart.firstsplit <- rpart(class ~ x1 + x2
, data = train_df
, method = "class"
, parms = list(split = "gini")
, control = rpart.control(minsplit = 2
, minbucket = 1
, maxdepth = 1
, cp = 0
, xval = 0
, maxcompete = 0
)
)
rpart.plot(cart.firstsplit)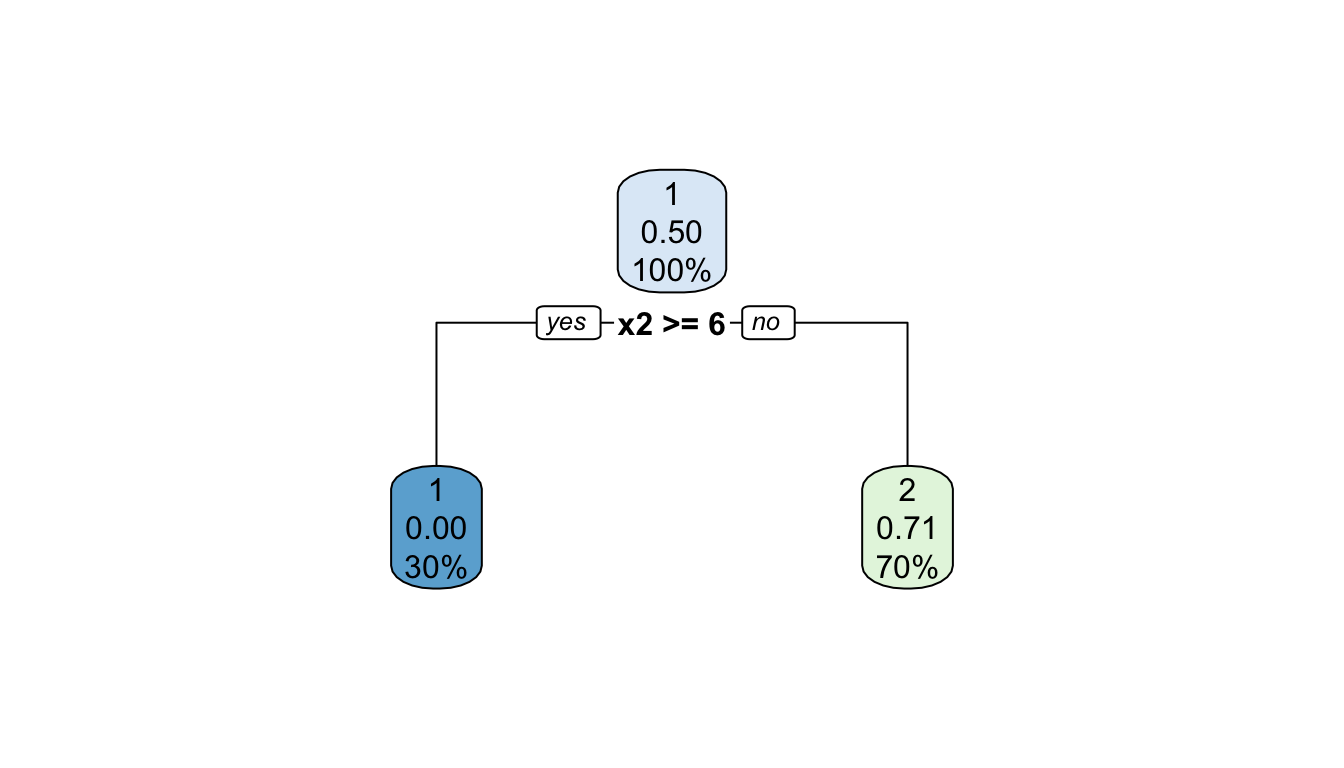
Figure 8.2: 뿌리노드 분지
또한 분지 결과 트리는 Table 8.2와 같이 frame이라는 이름의 data frame에 설명된다. 각 행 앞의 번호는 노드 인덱스 \(t\)를 나타내며, 각 열에 대한 설명은 아래와 같다.
- var: 노드 \(t\)를 분지하는 데 이용된 변수. 값이 <leaf>인 경우에는 노드 \(t\)가 최종 노드임을 나타낸다.
- n: 노드 내 객체 수 \(N(t)\)
- wt: 가중치 적용 후 객체 수 (추후 appendix에서 설명)
- dev: 오분류 객체 수
- yval: 노드 \(t\)를 대표하는 범주
- complexity: 노드 \(t\)에서 추가로 분지할 때 감소하는 relative error값; 본 분류트리 예제에서 error는 오분류율이며, 뿌리 노드의 relative error값을 1으로 한다.
| var | n | wt | dev | yval | complexity | ncompete | nsurrogate | |
|---|---|---|---|---|---|---|---|---|
| 1 | x2 | 10 | 10 | 5 | 1 | 0.6 | 0 | 0 |
| 2 | <leaf> | 3 | 3 | 0 | 1 | 0.0 | 0 | 0 |
| 3 | <leaf> | 7 | 7 | 2 | 2 | 0.0 | 0 | 0 |
또한 frame에는 트리 내 각 노드에 속한 객체와 범주에 대한 정보를 나타내는 yval2라는 행렬이 Table 8.3와 같이 존재한다. 실제 yval2의 열의 개수는 전체 학습 대상 범주 수에 따라 달라지며, 본 예는 이분 분류 트리(범주개수 = 2)에 해당하는 열 구성을 보여준다. 각 행 앞의 번호는 노드 인덱스 \(t\)를 나타내며, 각 열에 대한 설명은 아래와 같다.
- 열1: 노드 \(t\)에서의 최적 추정 범주 \(j^*\)
- 열2: 노드 \(t\) 내 범주 class=1 객체 수 \(N_1(t)\)
- 열3: 노드 \(t\) 내 범주 class=2 객체 수 \(N_2(t)\)
- 열4: 노드 \(t\) 내 범주 class=1 관측 확률 \(p(1|t) \approx \tfrac{N_1(t)}{N(t)}\)
- 열5: 노드 \(t\) 내 범주 class=2 관측 확률 \(p(2|t) \approx \tfrac{N_2(t)}{N(t)}\)
- nodeprob: 노드 \(t\) 확률 \(p(t) \approx \tfrac{N(t)}{N}\)
## Warning: Setting row names on a tibble is deprecated.| 열1 | 열2 | 열3 | 열4 | 열5 | nodeprob | |
|---|---|---|---|---|---|---|
| 1 | 1 | 5 | 5 | 0.50 | 0.50 | 1.0 |
| 2 | 1 | 3 | 0 | 1.00 | 0.00 | 0.3 |
| 3 | 2 | 2 | 5 | 0.29 | 0.71 | 0.7 |
위 CART 모델 데이터를 이용하여 트리의 불순도를 계산해보자.
우선 노드 상세 정보 행렬 yval2의 x번째 노드의 불순도(\(i(t)\))를 계산하는 함수 rpartNodeImpurity를 아래와 같이 구현한다.
rpartNodeImpurity <- function(x, yval2) {
node_vec <- yval2[x, ]
n.columns <- length(node_vec)
class.prob <- node_vec[((n.columns/2)+1):(n.columns-1)]
return(1 - sum(class.prob^2))
}CART tree 객체의 각 leaf node에 함수 rpartNodeImpurity를 적용하여 노드 불순도 \(i(t)\)를 계산한 뒤, 노드 확률 \(p(t)\)을 이용한 가중합을 통해 트리 불순도 \(I(T)\)를 계산하는 함수 rpartImpurity를 아래와 같이 구현한다.
rpartImpurity <- function(rpart.obj) {
leaf.nodes <- which(rpart.obj$frame$var=="<leaf>")
node.impurity <- sapply(leaf.nodes,
rpartNodeImpurity,
yval2 = rpart.obj$frame$yval2)
node.prob <- rpart.obj$frame$yval2[leaf.nodes, 'nodeprob']
return(sum(node.prob * node.impurity))
}위 함수를 이용하여 계산한 트리 Figure 8.1의 불순도는 0.29이다.
rpartImpurity(cart.firstsplit)## [1] 0.2857143분지를 추가할수록 불순도는 감소한다. 분지를 추가하기 위해서는 maxdepth라는 control parameter 값을 증가시키면 된다.
- maxdepth: 뿌리노드부터 임의의 최종노드에 도달하는 최대 가능 분지 수 (default=30)
maxdepth 파라미터의 값을 1부터 4까지 증가시키며 불순도의 변화를 살펴보자.
library(ggplot2)
tree.impurity <- sapply(c(1:4), function(depth) {
rpart(class ~ x1 + x2
, data = train_df
, method = "class"
, parms = list(split = "gini")
, control = rpart.control(minsplit = 2
, minbucket = 1
, maxdepth = depth
, cp = 0
, xval = 0
, maxcompete = 0)) %>%
rpartImpurity()
})
tibble(maxdepth=c(1:4), impurity=tree.impurity) %>%
ggplot(aes(x=maxdepth, y=impurity)) +
geom_line()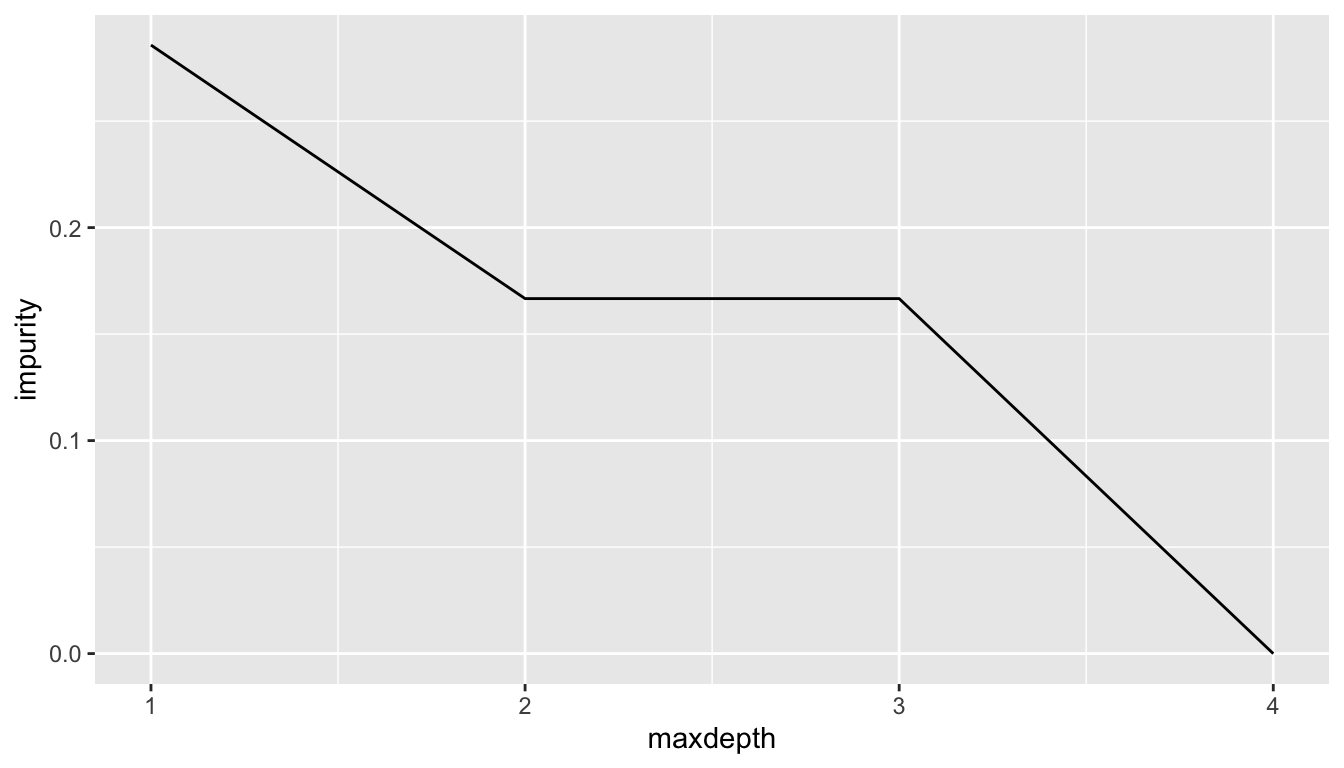
Figure 8.3: 파라미터 maxdepth값에 따른 트리불순도 변화
위 예에서, 트리의 분지가 증가함에 따라 불순도는 0.29, 0.17, 0.17, 0로 감소한다. maxdepth값이 3일 때 불순도가 감소하지 않는 이유는, 세 번째 분지 결과가 전체적인 오분류를 감소시키지 않아 rpart 함수가 해당 분지를 취소하기 때문이다. 여기에 작용하는 파라미터는 cp라는 control parameter이다.
- cp: 노드가 분지되기 위한 최소 relative error 감소치 (default = 0.01). 값이 0일 경우 최대트리를 생성한다.
위 예제에서는 cp값을 0으로 설정하여, 해당 분지가 트리 불순도를 감소시킨다 하더라도 전체 트리의 오분류를 감소시키는 데 기여하지 않는다면 시도하지 않도록 하였다.
8.4 가지치기 및 최종 트리 선정
8.4.1 가지치기
앞 장의 최대 트리 그림 8.1은 학습 데이터를 오분류 없이 완벽하게 분류하기 위해 복잡한 분류 구조를 형성하였다. 이러한 복잡한 분류 구조는 학습 데이터가 아닌 새로운 데이터에 대한 분류 정확도를 떨어뜨릴 수 있다. 이는 bias-variance tradeoff라 부르는 현상으로, 비단 분류트리 뿐 아니라 모든 데이터마이닝 방법에 일반적으로 적용된다.
분류 트리는 가지치기라는 방식을 통해, 분류 구조를 단순화함으로써 분류 트리가 새로운 데이터에도 정확한 분류를 제공하기를 추구한다. 가지치기란 트리 내 특정 내부노드를 기준으로 그 하위에 발생한 분지를 모두 제거하고, 해당 내부노드를 최종노드로 치환하는 방식이다.
| var | n | wt | dev | yval | complexity | ncompete | nsurrogate | |
|---|---|---|---|---|---|---|---|---|
| 1 | x2 | 10 | 10 | 5 | 1 | 0.6 | 0 | 0 |
| 2 | <leaf> | 3 | 3 | 0 | 1 | 0.0 | 0 | 0 |
| 3 | x1 | 7 | 7 | 2 | 2 | 0.2 | 0 | 0 |
| 6 | <leaf> | 1 | 1 | 0 | 1 | 0.0 | 0 | 0 |
| 7 | x2 | 6 | 6 | 1 | 2 | 0.1 | 0 | 0 |
| 14 | x1 | 2 | 2 | 1 | 1 | 0.1 | 0 | 0 |
| 28 | <leaf> | 1 | 1 | 0 | 1 | 0.0 | 0 | 0 |
| 29 | <leaf> | 1 | 1 | 0 | 2 | 0.0 | 0 | 0 |
| 15 | <leaf> | 4 | 4 | 0 | 2 | 0.0 | 0 | 0 |
Table 8.4에서 생성 가능한 가지치기는 최종 노드(var값이 <leaf>)가 아닌 모든 노드(1, 3, 7, 14)에서 가능하며, 함수 snip.rpart를 이용하여 가지치기 된 트리를 생성할 수 있다. 각 내부 노드에서 가지치기된 트리들은 아래와 같이 얻어진다.
internal.node.index <- rownames(cart.est$frame)[which(cart.est$frame$var != '<leaf>')] %>%
as.numeric()
snipped <- lapply(internal.node.index, function(x){snip.rpart(cart.est, x)})
n.trees <- length(snipped)
par(mfrow=c(2,2))
invisible(lapply(c(1:n.trees), function(x) {
rpart.plot(snipped[[x]])}
))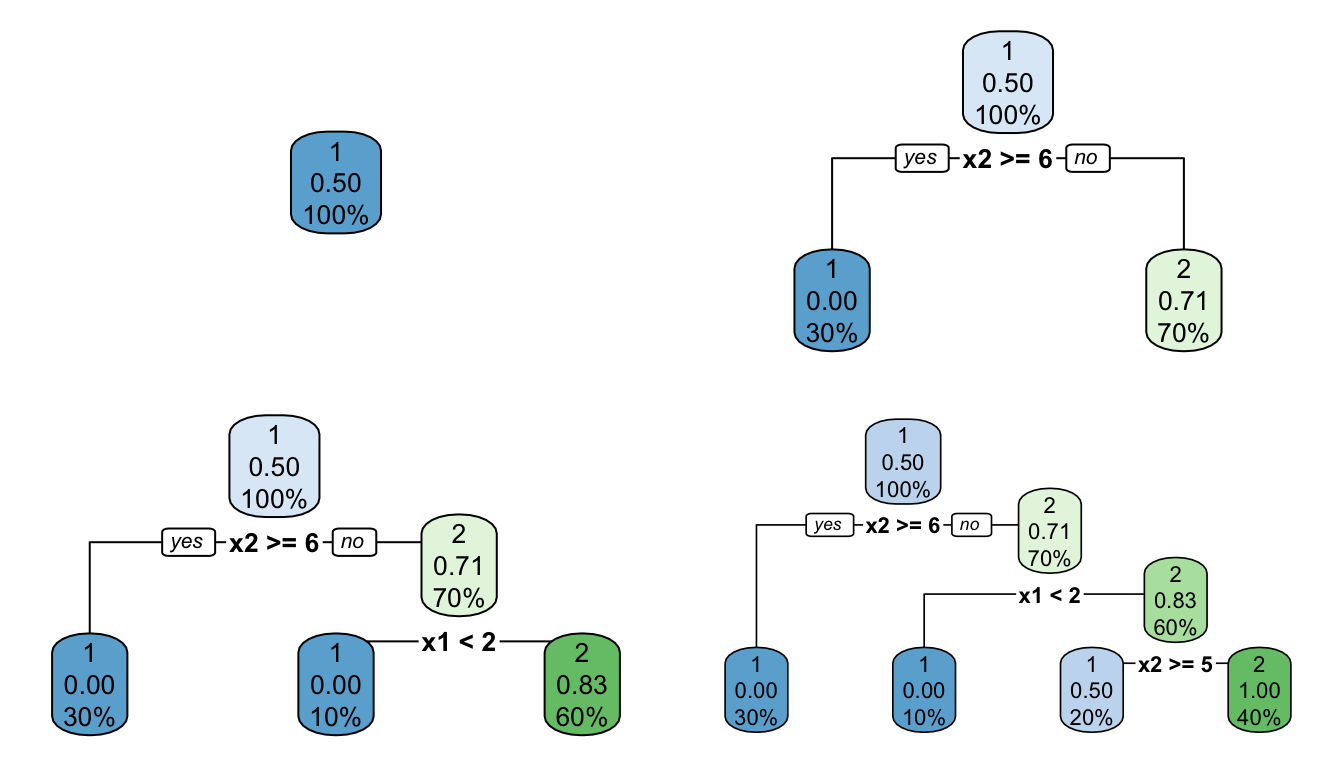
Figure 8.4: 각 내부노드 기준으로 가지치기된 트리
위 각 가지치기 후보 노드의 오분류 비용은 함수 nodeCost를 아래와 같이 구현하여 계산할 수 있다.
nodeCost <- function(node, tree) {
node_vec <- tree$frame$yval2[as.character(node) == row.names(tree$frame), ]
n.columns <- length(node_vec)
class.prob.max <- max(node_vec[((n.columns/2)+1):(n.columns-1)])
node.prob <- node_vec[n.columns]
node.misclassification.cost <- (1-class.prob.max)*node.prob
return(node.misclassification.cost)
}
tibble(
pruning_node = internal.node.index,
node_cost = sapply(internal.node.index, nodeCost, tree=cart.est)
) %>%
knitr::kable()| pruning_node | node_cost |
|---|---|
| 1 | 0.5 |
| 3 | 0.2 |
| 7 | 0.1 |
| 14 | 0.1 |
각 가지치기 노드에 해당하는 하부 트리의 오분류비용 및 복잡도를 구하기 위해 subtreeEval라는 함수를 아래와 같이 구현한다.
subtreeEval <- function(node, tree) {
snipped <- snip.rpart(tree, node)$frame
leaf.nodes <- setdiff(rownames(tree$frame[tree$frame$var=="<leaf>",]),
rownames(snipped)) %>%
as.numeric()
tibble(
pruning_node = node,
node.cost = nodeCost(node, tree),
subtree.cost = sapply(leaf.nodes, nodeCost, tree=tree) %>% sum(),
subtree.size = length(leaf.nodes)
) %>%
mutate(alpha = (node.cost - subtree.cost) / (subtree.size - 1))
}각 노드에 대하여 알파값을 다음과 같이 계산할 수 있다.
df.cost <- lapply(internal.node.index, subtreeEval, tree=cart.est) %>%
bind_rows()| pruning_node | node.cost | subtree.cost | subtree.size | alpha |
|---|---|---|---|---|
| 1 | 0.5 | 0 | 5 | 0.12 |
| 3 | 0.2 | 0 | 4 | 0.07 |
| 7 | 0.1 | 0 | 3 | 0.05 |
| 14 | 0.1 | 0 | 2 | 0.10 |
위 Table 8.5 에서 최소 알파값에 해당하는 노드 7에서 가지치기를 한다.
pruned.tree.1 <- snip.rpart(cart.est,
df.cost$pruning_node[which.min(df.cost$alpha)])
rpart.plot(pruned.tree.1)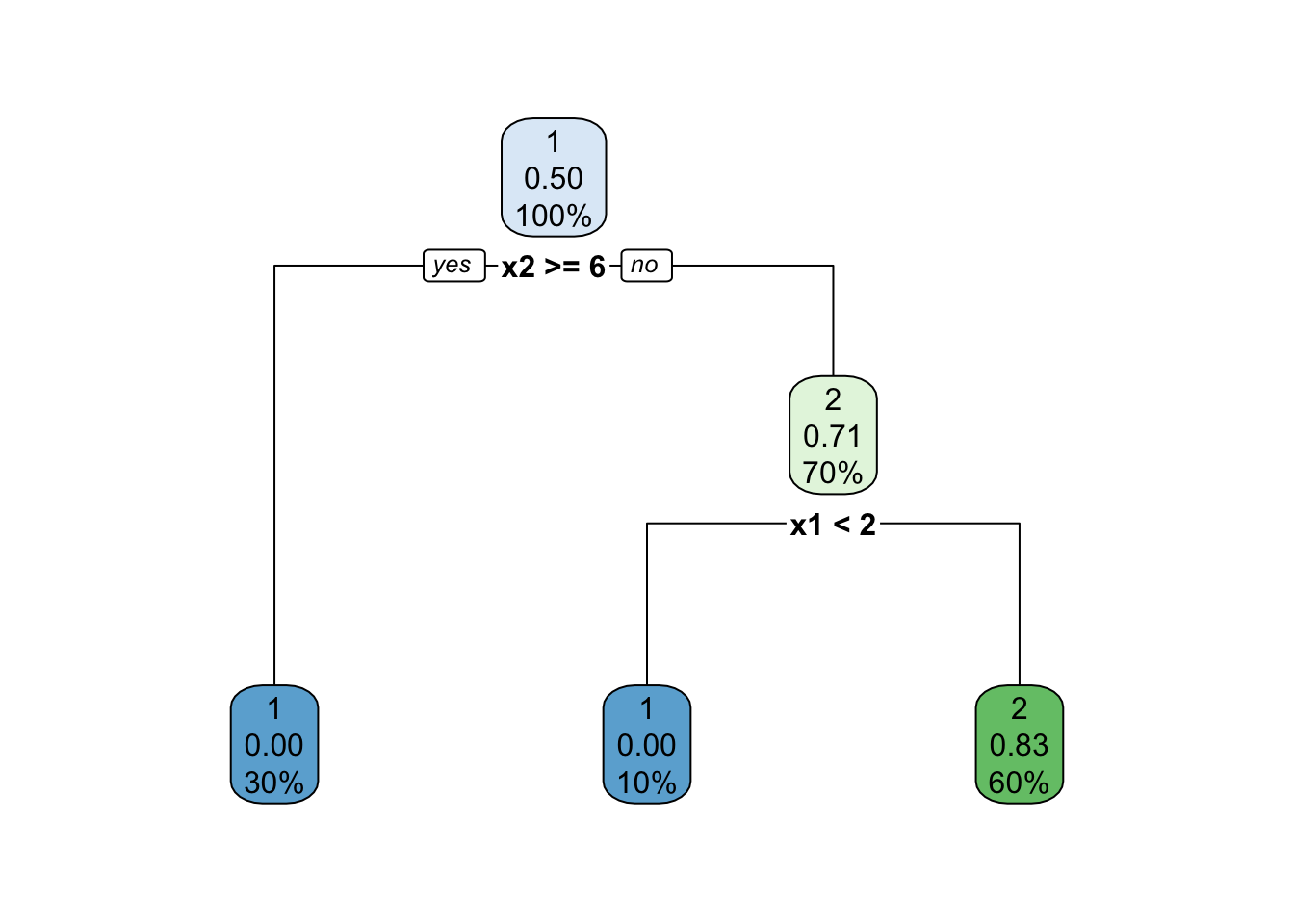
Figure 8.5: 1단계 가지치기 결과
가지치기로 형성된 트리에서 다시 각 가지치기 노드의 오분류비용, 복잡도 및 알파값을 구한다.
df.cost <- rownames(pruned.tree.1$frame)[pruned.tree.1$frame$var!="<leaf>"] %>%
as.numeric() %>%
lapply(subtreeEval, tree=pruned.tree.1) %>%
bind_rows()
knitr::kable(df.cost)| pruning_node | node.cost | subtree.cost | subtree.size | alpha |
|---|---|---|---|---|
| 1 | 0.5 | 0.1 | 3 | 0.2 |
| 3 | 0.2 | 0.1 | 2 | 0.1 |
위 결과에서 다시 최소 알파값에 해당하는 노드 3에서 가지치기를 하면 아래와 같은 트리가 형성된다.
pruned.tree.2 <- snip.rpart(pruned.tree.1,
df.cost$pruning_node[which.min(df.cost$alpha)])
rpart.plot(pruned.tree.2)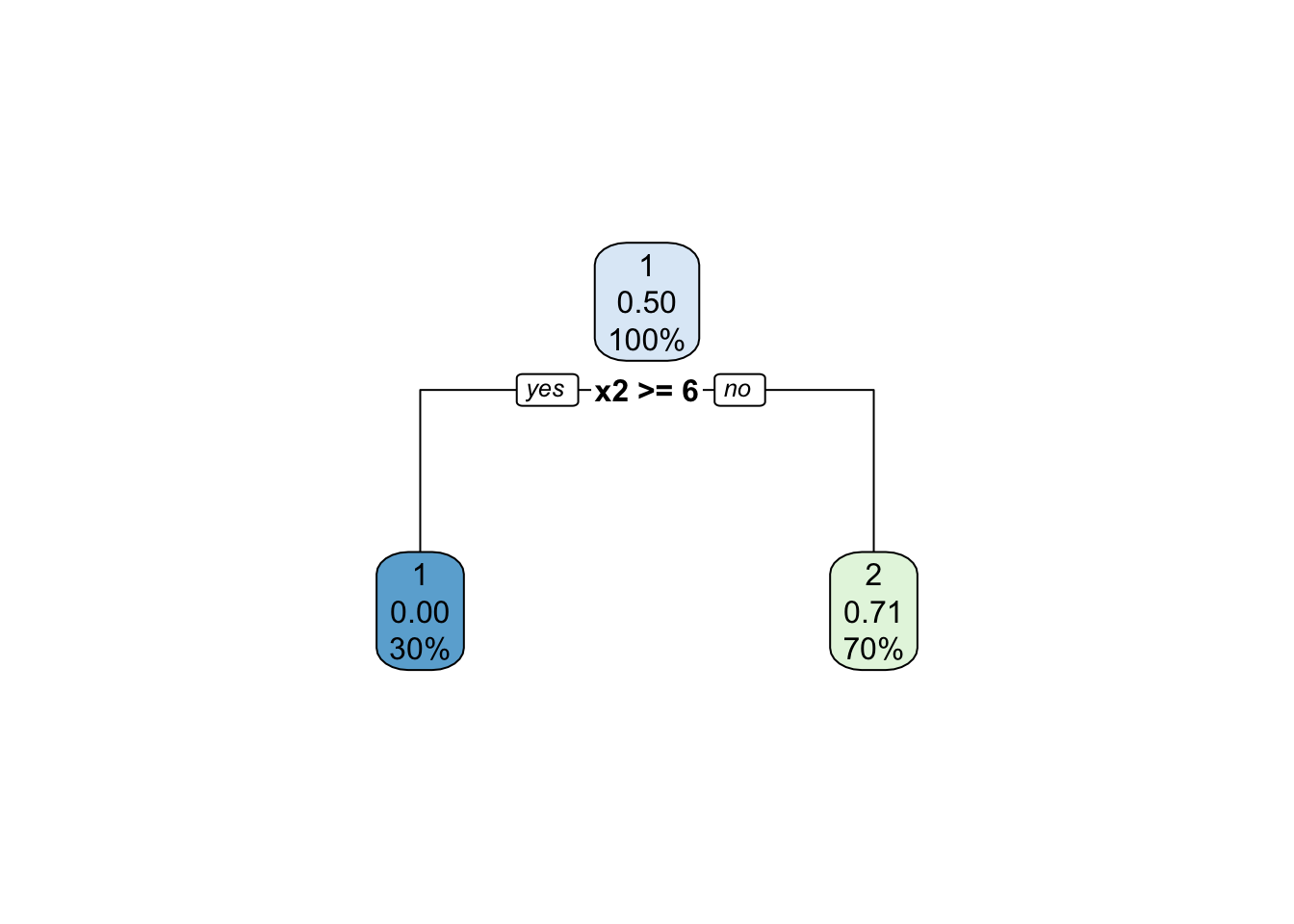
Figure 8.6: 2단계 가지치기 결과
8.4.2 최적 트리의 선정
위 가지치기 과정에서 얻는 가지친 트리들이 최종 트리의 후보가 되며, 이 중 테스트 표본에 대한 오분류율이 가장 작은 트리를 최적 트리로 선정하게 된다.
트리를 학습할 때 사용된 학습데이터 Table 8.1 외에, Table 8.6과 같은 6개의 테스트 데이터가 있다고 하자.
test_df <- tibble(
x1 = c(1,0,3,4,2,1),
x2 = c(5,5,4,3,7,4),
class = factor(c(1,1,2,2,1,2), levels=c(1,2))
)| x1 | x2 | class |
|---|---|---|
| 1 | 5 | 1 |
| 0 | 5 | 1 |
| 3 | 4 | 2 |
| 4 | 3 | 2 |
| 2 | 7 | 1 |
| 1 | 4 | 2 |
테스트 데이터에 위에서 학습된 세 개의 트리, 즉 최대 트리 cart.est와 두 개의 가지치기 트리 pruned.tree.1 & pruned.tree.2를 적용하여 각 트리가 각각의 테스트 데이터를 어떻게 분류하는지 살펴보자.
test_pred <- test_df %>%
bind_cols(
pred_maxtree = predict(cart.est, test_df, type="class"),
pred_prune1 = predict(pruned.tree.1, test_df, type="class"),
pred_prune2 = predict(pruned.tree.2, test_df, type="class")
)| x1 | x2 | class | pred_maxtree | pred_prune1 | pred_prune2 |
|---|---|---|---|---|---|
| 1 | 5 | 1 | 1 | 1 | 2 |
| 0 | 5 | 1 | 1 | 1 | 2 |
| 3 | 4 | 2 | 2 | 2 | 2 |
| 4 | 3 | 2 | 2 | 2 | 2 |
| 2 | 7 | 1 | 1 | 1 | 1 |
| 1 | 4 | 2 | 1 | 1 | 2 |
결과 Table 8.7에서 최대트리가 오분류한 테스트 표본은 1개, 첫번째 가지치기 트리가 오분류한 테스트 표본은 1개, 그리고 두 번째 가지치기 트리가 오분류한 테스트 표본은 2개이다.
위 결과를 토대로, 최적의 트리를 선정하는 과정은 아래와 같다.
- 각각의 트리에 의해 오분류된 테스트 표본의 개수를 전체 테스트 표본의 개수로 나누어 오분류율 \(R^{ts}\)를 구한다.
- 테스트 표본 수를 \(n_{test}\)라 할 때, 오분류의 표준편차를 아래와 같이 계산한다. \[SE = \sqrt{\frac{R^{ts}(1 - R^{ts})}{n_{test}}}\]
- 1에서 구한 오분류율에 2에서 구한 표준편차를 더하여 \(R^{ts} + SE\)를 각 트리의 평가척도로 계산한다. 후보 트리들 중 해당 평가척도가 가장 작은 트리를 최종 트리로 선정한다.
test.summary <- test_pred %>%
summarize(n.test = n(),
cart.est = sum(pred_maxtree != class) / n.test,
pruned.tree.1 = sum(pred_prune1 != class) / n.test,
pruned.tree.2 = sum(pred_prune2 != class) / n.test) %>%
gather("tree","R.ts",-n.test) %>%
mutate(SE = sqrt((R.ts*(1 - R.ts))/n.test),
score = R.ts + SE) %>%
select(-n.test)| 트리 | 오분류율(\(R^{ts}\)) | 표준편차(\(SE\)) | 척도(\(R^{ts} + SE\)) |
|---|---|---|---|
| cart.est | 0.17 | 0.15 | 0.32 |
| pruned.tree.1 | 0.17 | 0.15 | 0.32 |
| pruned.tree.2 | 0.33 | 0.19 | 0.53 |
위 결과, 최적 트리는 최대 트리 혹은 첫 번째 가지치기 트리가 된다.
위 절차를 임의의 데이터에 대해 수행하는 함수를 구현해보자.
rpart_learn <- function(formula, train_df, test_df) {
# 최대 트리 생성
max_tree <- rpart(formula
, data = train_df
, method = "class"
, parms = list(split = "gini")
, control = rpart.control(minsplit = 2
, minbucket = 1
, cp = 0
, xval = 0
, maxcompete = 0
)
)
# 가지치기
curr_tree <- list()
k <- 1
curr_tree[[k]] <- max_tree
while(dim(curr_tree[[k]]$frame)[1] > 1) {
internal.node.index <- rownames(curr_tree[[k]]$frame)[which(curr_tree[[k]]$frame$var != '<leaf>')] %>%
as.numeric()
df.cost <- lapply(internal.node.index, subtreeEval, tree=curr_tree[[k]]) %>% bind_rows()
curr_tree[[k + 1]] <- snip.rpart(curr_tree[[k]],
df.cost$pruning_node[which.min(df.cost$alpha)])
k <- k + 1
}
# 최적 가지치기 트리 선정
n.test <- dim(test_df)[1]
R.ts <- lapply(curr_tree, function(x) {
sum(predict(x, test_df, type="class") != test_df$class) / n.test
}) %>% unlist()
score <- R.ts + sqrt((R.ts*(1 - R.ts))/n.test)
return(curr_tree[[max(which(score == min(score)))]])
}
optimal_tree <- rpart_learn(class ~ x1 + x2, train_df, test_df)
rpart.plot(optimal_tree)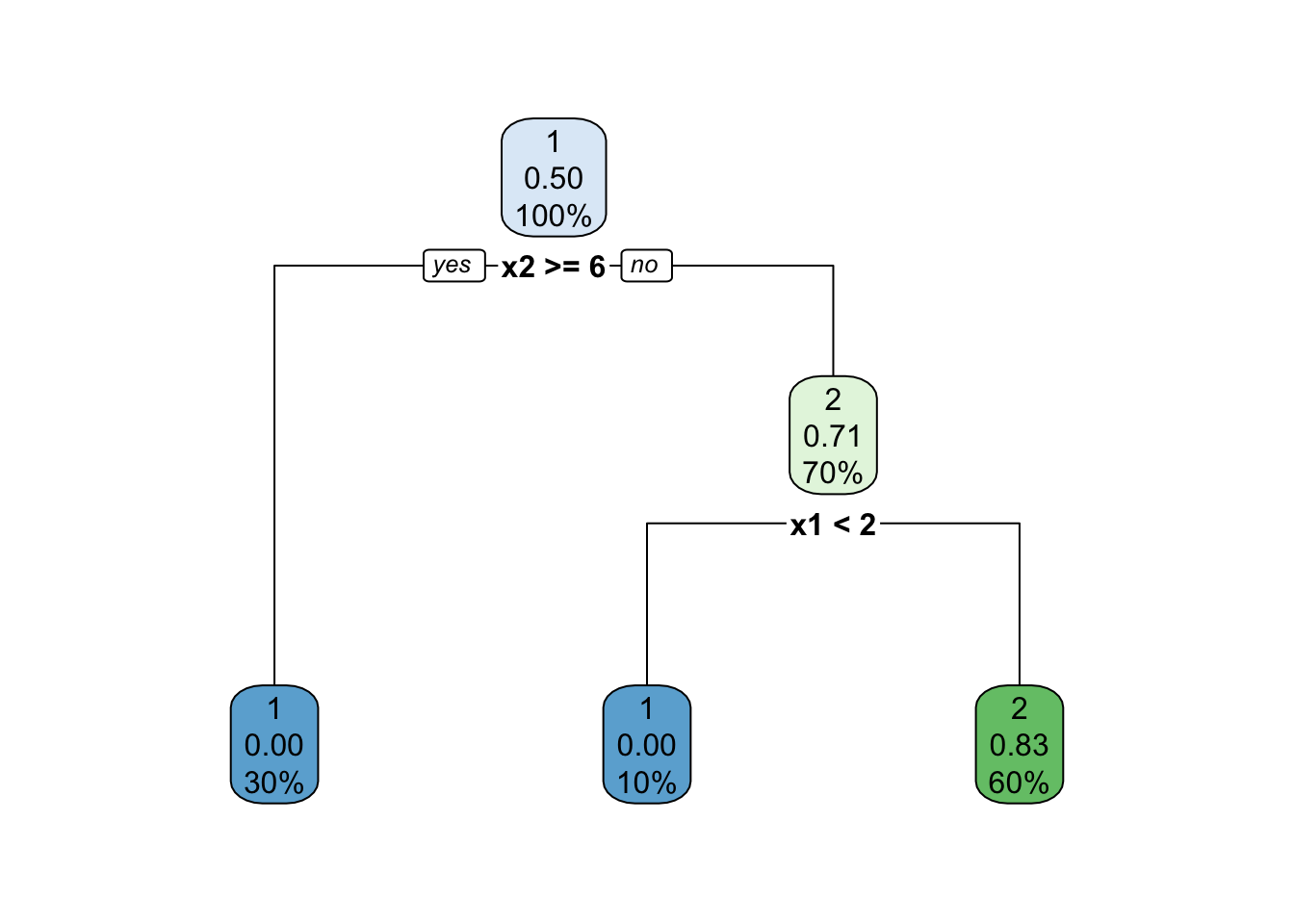
8.5 R패키지 내 분류 트리 방법
앞 장에서는 rpart의 결과를 이용하여 교재 8.2 - 8.3장의 예제를 재현해보았다. 실제로 rpart 내부의 기본 트리 방법은 교재의 예제와는 다소 다른 부분이 있다. 이 장에서는 실제 rpart 패키지의 분류 트리 방법에 대해 알아본다.
8.5.1 트리 확장
트리 내 임의의 노드 \(t\)에 대한 불순도는 아래와 같이 정의된다. \[i(t) = \sum_{j=1}^{J} f\left(p(j|t)\right)\] 여기에서 \(p(j|t)\)는 노드 \(t\) 내 전체 샘플 \(N(t)\) 중 범주 \(j\)의 샘플 \(N_j(t)\)의 비율로 추정된다. \[p(j|t) \approx \frac{N_j(t)}{N(t)}\] 또한 함수 \(f\)는 concave 함수로, \(f(0) = f(1) = 0\)의 조건을 만족시켜야 한다. rpart 에서 설정할 수 있는 함수 \(f\)의 종류에 대해서는 아래에서 좀 더 자세히 살펴보기로 한다.
트리 내 임의의 노드 \(t\)가 분지규칙 \(s\)에 따라 두 개의 노드 \(t_L\)과 \(t_R\)로 분지된다고 할 때, 불순도의 감소량은 아래와 같이 계산된다.
\[\begin{eqnarray} \Delta I(s,t) &=& I(t) - I(t_L) - I(t_R)\\ &=& p(t)i(t) - p(t_L)i(t_L) - p(t_R)i(t_R) \end{eqnarray}\]
rpart는 위 \(\Delta I(s,t)\)값이 최대가 되는 분지 기준 \(s^*\)를 찾아 노드 \(t\)를 분지하여 트리를 확장하고, 확장된 트리의 최종 노드에서 다시 최적 분지를 찾는 과정을 반복한다.
8.5.1.1 분지 함수
함수 rpart 사용 시 parms 파라미터에 split 값으로 분지 방법을 설정할 수 있다.
- Gini index (parms=list(split=‘gini’)) 교재의 예제에 사용된 방법으로, 우선 아래와 같은 함수 \(f\)를 사용한다. \[f(p) = p(1-p)\]
- information index (parms=list(split=‘information’)) 교재에 엔트로피 지수(Entropy index)로 설명된 지수로, 아래와 같은 함수를 사용한다. \[f(p) = -p\log(p)\]
- user-defined function 사용자가 임의로 함수를 정의하여 사용할 수 있다. 본 장에서는 자세한 설명은 생략한다.
8.5.2 가지치기
임의의 노드 \(t\)에 대한 위험도(오분류 비용의 기대치)는 아래와 같이 계산된다. \[r(t) = \sum_{j \neq \tau(t)} p(j|t)C\left(\tau(t)|j\right)\] 여기에서 함수 \(C(i|j)\)는 범주 \(j\)에 속하는 객체를 범주 \(i\)로 분류할 때의 오분류 비용이며, \(\tau(t)\)는 노드 \(t\) 내의 오분류 비용을 최소화하도록 노드 \(t\)에 지정된 범주값이다.
rpart의 오분류 비용 \(C(i|j)\)의 기본값은 \[C(i|j) = \begin{cases} 1, & \text{ if } i \neq j\\ 0, & \text{ if } i = j \end{cases} \] 으로 설정되어 있으며, parms 파라미터에 loss 값으로 오분류 비용 \(C(i|j)\)를 재설정할 수 있다. 본 장에서는 기본값을 사용하도록 하자.
\(A(T)\)를 트리 \(T\)의 최종 노드의 집합이라 정의하고, 트리의 최종 노드의 개수를 \(|T|\)라 할 때, 트리 \(T\)의 위험도 \(R(T)\)는 아래와 같이 정의된다. \[R(T) = \sum_{t \in A(T)} p(t)r(t)\]
복잡도 계수(complexity parameter) \(\alpha \in [0, \infty)\)를 이용하여, 트리의 비용-복합도 척도를 다음과 같이 정의한다. \[R_\alpha(T) = R(T) + \alpha|T|\] 이 때, 임의의 계수 \(\alpha\)에 대해 비용 \(R_\alpha(T)\)가 최소가 되게하는 가지치기 트리를 \(T_\alpha\)라 하면, 아래와 같은 관계들이 성립한다.
- \(T_0\): 최대 트리
- \(T_\infty\): 뿌리 노드 트리 (분지 없음)
- \(\alpha > \beta\)일 때, \(T_\alpha\)는 \(T_\beta\)와 동일하거나 혹은 \(T_\beta\)에서 가지치기된 트리이다.
8.5.3 파라미터값 결정
함수 rpart를 사용할 때 여러가지 사용자 정의 파라미터값을 설정할 수 있으며, 그 파라미터 값에 따라 생성되는 트리의 결과가 달라진다. 대표적인 파라미터 값으로는 아래와 같은 것들이 있다.
- minsplit: 분지를 시도하기 위해 필요한 노드 내 최소 관측객체 수 (default=20)
- cp: 노드가 분지되기 위한 최소 relative error 감소치 (default = 0.01). 값이 0일 경우 최대트리를 생성한다.
- maxdepth: 뿌리노드부터 임의의 최종노드에 도달하는 최대 가능 분지 수 (default=30)